介绍语义集成融汇概念,语义网技术在集成融汇中的作用;总结语义集成融汇关键技术,包括语义化数据描述技术、基于语义的协议规范、基于本体的融汇推理技术三个方面;分析国外主要研究项目,包括KC3 Browser、Bio2RDF、SBWS和Semantic REST等;最后指出如何推动其发展。
This paper first introduces the concept of Semantic Mashup and the role of Semantic Web in the Mashup process. Then it summarizes the crucial technologies of Semantic Mashup, such as semantic data describing technology, semantic protocol specifications, Ontology-based Mashup reasoning technology. In addition, the paper analyzes some oversea on-going research projects, specifically for KC3 Browser, Bio2RDF, SBWS and Semantic REST. And finally, it gives some advice on how to promote the development of Semantic Mashup.
当前,集成融汇(Mashup)的主要方法包括:数据类型融汇法(Datatype-based)、模式融汇法(Model-based)、大纲融汇法(Schema-based)、实例融汇法(Instance-based)等,这些方法虽然支持异构资源的融汇,但在可用性、灵活性、兼容性、表现性以及支持机器自动融汇方面存在一定的局限性,而语义集成融汇法(Semantics-based)则代表未来的发展方向[ 1]。语义集成融汇(Semantic Mashup)是将语义网技术与Mashup技术相结合,通过对集成融汇的资源对象、服务对象进行语义描述,并建立对象间的语义关系,支持不同来源、不同类型资源间的语义映射、推理,实现复杂语义层面的信息对象的融合、汇集。
语义网技术在Mashup中可以起到特殊作用[ 2]:
(1)通过在网上发布RDF格式数据,不但可以克服异构数据集成的障碍,而且可以支持数据集之间的关联整合,支持利用语义网浏览器(比如Piggy Bank)浏览关联数据项。RDF链接可以被语义网搜索引擎所采集和标引,例如Sindice.com支持采集数据的复杂查询,由于查询结果是结构化的数据,这些结果可以很方便地被其他应用集成融汇。
(2)利用语义网的链接、推理技术,可以创建本体和数据实体之间的映射,进而支持不同领域数据的语义融汇。同时,采用语义网技术对资源进行标注,使用基于内容与注释的分离方式对服务描述和服务请求中的领域概念进行扩展,有助于服务发现及工作流重组。
(1)基于RDF、URI的数据组织
RDF是用于描述网络资源的标记语言,通过RDF用户可以使用自定义的词汇表描述任何资源,由于使用的是结构化的XML数据,搜索引擎可以理解元数据的精确含义,使得搜索变得更加智能、准确。同时,网络上任何可用的资源都可以通过通用资源标识符URI进行定位。基于RDF、URI等技术,在组织、发布Web数据的同时可以建立不同数据源之间的数据链接,进而支持对数据、信息、知识进行语义揭示、共享和链接操作。目前,很多组织机构都在积极推动语义化数据组织建设,例如W3C设立了Linked Open Data项目[ 3],其目标就是采用RDF、URI等技术对开放网络资源进行重新组织,发布语义化、链接化的数据集,支持语义层面的开放调用与集成融汇。该项目已经发布了100多个语义化数据集,包括DBpedia[ 4],WordNet[ 5],RDF Book Mashup[ 6]等。其中,DBpedia是从Wikipedia抽取结构化信息,利用RDF进行描述并建立对象间URI链接。 每个三元组由主、谓、宾三部分组成,每个部分可以是文字或URI,每个URI都意味着一个指向新链接资源的描述。这种描述通常包含新的指向其他URI的RDF链接。RDF链接使得利用语义网浏览器可以浏览一个数据源中的数据项与其他数据源中相关的数据。RDF链接可以被语义网搜索引擎采集到,在这些采集的数据上做更复杂的检索,而且检索结果也是结构化的数据,可以被其他的应用程序所集成。
(2)基于RDFa的语义标注
RDFa是直接在XHTML内包含RDF数据的一种机制。RDFa使用XHTML元素和属性的集合,使网页能够包含任意数量和任意复杂性的机器可读的语义数据,并显示标准的XHTML 内容[ 7]。如例1所示,通过在HTML标签中添加有关的描述属性,指明该标签的语义。本例中通过在<a>标签中增加属性:rel="license",表明该链接的语义是版权协议;在<h2>标签中增加属性:property="dc:title",表明该内容为标题;在&图片h3>标签中增加属性:property="dc:creator",表明该标签内容是作者。利用这些语义标记,第三方系统就可以快捷、准确地从网页中抽取相关信息项,进而完成与其他资源的语义融汇。
例1:
A Creative Commons License
<h2 property="dc:title">The trouble with Bob</h2>
<h3 property="dc:creator">Alice
……
(3)基于Microformats的语义标注
与RDFa类似,Microformats是在HTML元素的类、属性中嵌入轻量级的语义标签,实现语义标注目标。Microformats包括基本微格式(Elemental Microformat)和复合微格式(Compound Microformat)[ 8]。基本微格式是解决单一问题的最小解决方案,采用XHTML支持的Rel、Rev、Class等属性定义具有语义的属性集,嵌入到网页文件中直接使用,或者作为复合微格式的基本组成要素。复合微格式由基本微格式和标准的XHTML 元素组成,解决描述复合数据类型现存标准方案与XHTML 之间的准确转换问题。基于微格式标注的网页数据不但直接支持语义融汇,通过利用GRDDL(Gleaning Resource Descriptions from Dialects of Languages)、XSLT等技术可以将其转换为RDF格式数据,支持基于语义网标准的融汇操作。例如英国爱丁堡大学Harry Halpin专门研究Microformats转换,试图为每种Microformats给出一种同类型的RDF模型(见表1)[ 9]。当前,利用Microformats和GRDDL开发社交网络的研究成为语义Mashup在社交网络中的一种典型应用。
| 表1 Microformats与RDF模型的映射 |
(4)直接从HTML网页中抽取语义内容
直接从HTML网页中抽取语义内容是语义融汇技术研究的重要方面。MIT开发了Firefox插件Piggy Bank[ 10],目标是支持用户从网页抽取信息实现语义融汇。当网页中不包含RDF数据时,Piggy Bank可以调用Screen Scrapers把网页信息重新构造成语义网格式,以RDF格式进行保存,供以后检索和与其他人共享。MIT还开发了Solvent[ 11]工具以帮助编写Scraper。
基于Web Service的开放API是支持集成融汇的关键技术。为实现从Web Service到Semantic Service的转变,W3C提出基于SAWSDL(Semantic Annotations for WSDL and XML Schema)的WSDL语义注释方案,支持SOAP的语义化信息交换[ 12];利用RDFa和GRDDL支持REST语义标注,实现基于REST的语义融汇[ 13]。
(1)SAWSDL
SAWSDL试图通过从WSDL和XSD元素中引用语义模型的方法来填补Web服务和语义网的鸿沟,这些语义模型主要指RDF和OWL,目的是将本体对象的URI作为属性嵌入在WSDL中。SAWSDL采用两种标注方法:
① 利用扩展属性modelReference,指定WSDL或XML Schema组件与某些语义模型中概念之间的关联,标注WSDL接口、操作和故障,以及XML Schema类型定义、元素声明和属性声明。
② 利用扩展属性liftingSchemaMapping和loweringSchemaMapping,指定语义数据和XML之间的映射,嵌入到XML Schema元素声明和类型定义中。
modelReference用于直接引用语义模型中的概念,如果一个组件或元素不能被直接引用,就可使用liftingSchemaMapping和 loweringSchemaMapping来指明数据映射转换,前者用于XML到语义数据的转换,后者用于语义数据到XML的转换。2007年8月28日,SAWSDL成为W3C的推荐标准。
(2)SA-REST
借鉴WSDL-S和SAWSDL中的相关思想,SA-REST通过使用RDFa和GRDDL添加和提取注释,实现为REST服务增加语义信息的目标。与SAWSDL不同, SA-REST注释必须添加到由HTML组成的网页中的服务。SA-REST采用RDFa将RDF三元组嵌入到XML、HTML或者XHTML文档中,支持URI和命名空间。采用GRDDL实现XML、XHTML格式到语义网数据的转换,它可以利用类似 XSLT 的程序从 XHTML 和 XML 的内容中提取声明,也可以从HTML相关的微格式中抽取RDF/XML。作为轻量级融汇协议,语义REST代表未来发展方向。
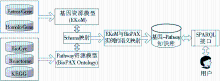
语义化访问协议、数据描述规范为数据的获取、融合提供了基本支持,但要支持计算机自动化数据分析、过滤、组合等操作,还需要建立相应的语义推理机制。当前研究主要采用本体对数据源进行描述,并利用本体之间的推理机制实现不同来源数据的语义融合。例如,在美国国立卫生研究院和美国国立医学图书馆项目SMGP(Semantic Mashup of Gene and Pathway)[ 14]中,研究人员采用本体映射方法实现尼古丁依赖性研究相关资源的语义融汇。尼古丁依赖性研究主要涉及两种基因资源(Entrez Gene和Homolo Gene)和三种Pathway资源(KEGG、Reactome和BioCyc)。如图1所示,项目组采用OWL为基因资源创建了知识模型EKoM(Entrez Knowledge Model),然后把该模型与现有的关于Pathway 资源的BioPAX本体集成。
在EKoM和BioPAX集成过程中,研究人员发现三个潜在的相似概念:Pathway、Protein、Interaction。通过选择在EKoM中重用概念“Pathway” 和“Protein”实现两类资源之间的语义关联。经过以上本体集成,系统支持用户通过RDF 查询语言SPARQL对相关资源的语义查询,并利用两个本体之间的映射关系发现相关的基因信息和Pathway信息。与SMGP类似的项目还有加拿大卡尔顿大学的yOWL[ 15]。
KC3 Browser[ 16]是由日本的情报通信研究机构(National Institute of Information and Communications Technology)开发的一个语义融汇浏览器。它根据用户的环境,如兴趣、目的、计算环境等,对无缝的知识存取实现自由链接浏览,并带有各种可视化组件。KC3 Browser是一个三层架构,最底层是Web Service层,它提供各种信息源,支持信息抽取、知识挖掘。中间是Mashup层,负责把内容和服务集成起来作为知识集,又称为K-Workspace,由语义资源、可视化资源、个人和社会资源、内容资源组成。K-Workspace中所有的内容和图表对象采用基于知识模型元数据标注,内部或外部的语义链接称为K-Links,由链接生成器产生。最顶层是Presentation层,支持显示、发表、共享、重用K-Workspace等功能。K-Workspace包含一个或多个K-Gadgets,它提供简单的功能单元,通过协同工作支持基于用户上下文信息的语义Mashup。目前,KC3 Browser被应用到一个应对东亚和欧盟自然灾害的风险情报项目中,提供预警、时间轴、地图、文档、用户概要、专家信息、三维地图、三维语义空间、风险分析图表等K-Gadgets。实验表明采用语义融汇和自由链接法超越了网络上时间、空间和领域界限,从而提高了系统的知识分析、共享和重用能力。
Bio2RDF[ 17]是由Genome Canada/Genome Quebec资助的生物信息学知识融汇项目。
Bio2RDF利用三元组库Sesame、本体编辑工具Protégé、语义浏览器Piggy Bank、RDF图观察器Welkin和LSID浏览器等工具,实现了生物信息学数据的集成融汇。具体过程分三步:
(1) 建立一个关于不同数据提供者的命名空间的列表,使得创建标准URI成为可能。
(2) 分析数据源,并用RDF模型表示。
(3) 建立一个RDFizer,把KEGG、PDB、MGI、HGNC和NCBI等数据源信息转换成RDF格式。
经过以上三步产生的RDF文档被存储在三元组库中,之后就可以采用相关软件工具对三元组库进行语义分析以及基于SeRQL的语义查询。Bio2RDF之所以选择RDF,是因为其图形匹配能力可为数据语义融汇提供强有力的支持,当两个RDF图之间存在某个URL相同时,这两个图即可合并成统一的数据结构。类似的还有Stephens主持的一个集成项目[ 18],同样采用RDF技术整合不同生物医学数据源,以支持药物发现。
美国BBN公司对语义网与Web Service的结合提出了两种解决方案,分别利用Web Service语义桥和Semantic REST方式实现语义集成融汇[ 19],目前该项目已经完成了一个结合两种方案的应用实例,实现了对Web2.0资源的分布式语义查询。其中,SBWS(Semantic Bridge for Web Service)通过在传统的SOAP和REST协议上增加语义标签,支持语义层次的服务组合。它封装了一套由WSDL或者WADL文档描述的Web Service操作,并为这些服务创建SPARQL查询端,SBWS通过分析SPARQL的SELECT或者CONSTRUCT决定Web Service操作的组合方式。SBWS利用WSDL文档和OWL-S文档查询SOAP类型的Web Service,基于SOAP服务由WSDL文档定义相关操作。对于REST服务没有对应的标准,SBWS选择来自于Sun Microsystem的WADL作为REST服务的详细说明。WADL被设计成WSDL的一种简单选择,用一种简单格式描述网络应用程序,同时定义了输入输出参数的格式,并通过对WADL文档自定义注释描述REST服务的语义。与SBWS不同,Semantic REST利用SPARQL和RDF定义约束和模型,从而实现直接从REST服务终端查询、检索、修改、删除、增加RDF数据的目标。Semantic REST操作面向两种类型的端点:类层和资源层。类层端点表示在远端服务器上的同一类资源,这些端点提供同类资源的创建和对同类资源的扩展查询。资源层端点表示某一个资源的URI,这些资源层的端点提供RDF格式的资源信息。
尽管目前已经出现了一些语义集成融汇的相关应用,但由于语义网技术的许多方面还不成熟,对于语义网与Mashup结合的研究也只是刚刚起步,所以为了推动语义集成融汇的发展也需要一些相应的机制,如鼓励服务提供者提供简单的REST API,可以在Web浏览器中被第三方使用;鼓励服务提供者以RDF格式发布数据,或者建立相应的系统把遗留数据以RDF格式导入到网络中,并且提供一个SPARQL终端,使得语义网的搜索引擎可以检索到相应的数据集;鼓励社区用户使用共享的语义术语标记内容等。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|