当前, 生物医学领域的信息急速膨胀, 各领域数据主要以机器无法进行语义识别的若干信息孤岛堆砌而成, 各个信息节点之间存在交流障碍。要将这些数据融合并对其进行推理以实现对现有数据的高效利用, 标准化的本体是必不可少的。然而由于本体异构, 大量的领域本体缺乏统一的语义表示和结构, 本体孤岛现象明显, 严重阻碍了数据的互操作与深度应用。为了提高数据的语义表达能力和开放互联能力, 美国国家生物医学本体中心(National Center for Biomedical Ontology, NCBO) 提出了数据共享项目来解决科学本体融合工具缺乏的现状。
美国国立医学图书馆开发的统一医学语言系统(UMLS) 是当今生物医学领域广泛使用的顶层本体, 它包括了100 多个领域本体。领域本体之间的映射是实现生物医学领域数据互操作的重要前提。本研究以 UMLS 包括的药事管理医学词典(The Medical Dictionary for Regulatory Activities, MedDRA) 与国际疾病分类(International Classfication of Diseases, ICD)为研究对象,探索基于NCBO的领域本体映射实现方法,并利用Java编程对半结构化XML结果的解析方法进行研究,从而得到可用于语义计算的结构化数据。通过对半结构化的结果进行解析,提高NCBO本体映射结果的可用性,并对解析结果进行了应用研究。本研究将为相关领域本体融合项目建设提供有益的借鉴。
生物医学领域的各类信息以指数形式不断增长, 包括实验室/病理学数据、电子病历数据、临床试验数据、患者病史数据、医学影像数据、微序列数据、蛋白质芯片数据、流式细胞术数据、基因类型/单核苷酸多态性(SNP) 数据。各领域数据以信息孤岛形式呈现, 多数的信息是无法利用机器进行语义识别的, 致使各个信息节点之间的交流存在障碍, 不利于生物医学研究与知识发现。尽管现存各种各样的标准, 相比之下, 本体在语义上是最完整的[ 1], 而且现有研究表明本体可以促进信息检索, 通过提供通用结构及术语对数据进行重复利用, 从而实现领域内、跨领域、跨专业、跨术语粒度的集成。因此标准化的本体是实现数据融合及高效利用的必要工具。在本体融合工具缺乏的现状下, NCBO提出了数据共享项目, 提供相应的本体服务, 实现科学本体的开放性、复用性和与其他科学本体的兼容性, 进而提高本体利用的效率[ 2]。
NCBO有6个核心组成部分,包括计算机科学与生物医学信息学研究、推进生物学项目与外部研究协作、基础设施、教育、传播和管理。目标是利用主要本体在网上提供可以语义互操作的生物医学知识和数据。生物医学信息学领域的研究者可以向NCBO提交本体, NCBO将本体存储在一个开放的生物医学本体库中, 即NCBO BioPortal, 然后用户可以浏览、搜索本体;上传新版本来对本体进行更新;实现本体可视化、对本体进行评论、评价或者分享他们的本体使用经验。
到目前为止, NCBO BioPortal集成了270多个本体。除了NCBO的本体, BioPortal提供的本体还包括美国国立医学图书馆人工集成的统一医学语言系统(UMLS) 与开放生物学和生物医学本体(OBO) , 提供的本体形式多样, 包括OWL、RDF(S) 、OBO和RRF(UMLS术语格式) 。
BioPortal提供的映射是不同本体之间术语的点对点映射(Point-to-Point Mapping) , 某些映射是人工定义的, 有些是利用各种映射算法自动创建的[ 3]。提供映射的资源包括三个:NCBO、UMLS和OBO。这三种资源的映射方式和关系如表1所示:
表1 NCBO本体映射资源、方式及关系

其中, “relatedMatch”指的是两个概念之间的相关映射;“closeMatch”指的是两个概念足够相似, 在某些信息检索应用中可以交互使用;“exactMatch”指的是两个概念之间高度相似, 可以在广泛的信息检索应用中交互使用。除了在NCBO、UMLS与OBO三个资源内部可以进行本体映射, 也可以实现三个资源间的本体映射。即NCBO不仅实现了基于顶层本体统控的本体映射, 也实现了异构本体之间的映射。
根据NCBO提供的本体映射类型, 映射服务大致可以分为两类:
(1) 不同本体之间的某些术语映射
这种映射局限于个体术语映射, 可以使用户了解不同本体中相关术语的使用情况。例如用户可以查找“ICD9CM”中的概念“心脏病”是否与MedDRA中的相关术语存在映射。这种映射的结果通常数量较小。
(2) 不同本体之间的整体术语映射
这种映射可以使用户了解不同本体之间的重复情况。例如用户可以查找MedDRA和ICD9CM之间的整体映射, 获得两个本体之间所有的映射结果。一般来说, 当两个本体间重复范围较大时, 这种映射的结果通常数量庞大。
NCBO本体映射方法包括通过网络界面互动操作, 以及通过代表性状态传输(Representational State Transfer, REST) 在线应用服务编程获取不同本体的映射信息[ 4]。网络界面互动操作简单易用, 但是对结果的限定不够精确, REST可以通过参数设定和编程获取用户最需要的结果。
REST是由Fielding[ 5]在他的博士论文中提出的一种架构思想, 是基于 SOAP 和 Web 服务描述语言(Web Services Description Language, WSDL) 的 Web 服务的更为简单的替代方法, 在 Web 领域已经得到了广泛的接受。REST的自描述、无状态、唯一标识等特性可以提供清晰、友好的应用程序编程接口(API) , 通过唯一的通用资源标识符(URI) 在网上为用户提供资源。并且, 目录结构式的URI具有层次结构, 它的根是单个路径, 从根开始分支的是公开服务的主要内容的子路径。作为网上获取资源的接口, 在使用中, URI 的结构简单、可预测且易于理解。
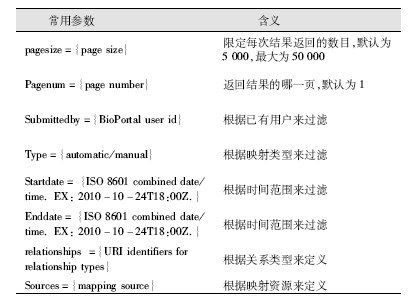
在NCBO中, 所有REST程序的前缀都是http://rest.bioontology.org/bioportal, 在前缀后面, 可以加上各种参数来进行限定, 常用的参数如表2所示:
表2 NCBO本体映射在线应用的常用参数
利用任何REST服务都需要使用一个“APIKey”, 因此首先要在http://bioportal.bioontology.org/login网页注册, 在“Account”处获得“APIKey”。使用NCBO的本体映射在线应用服务时, 在网页地址栏中输入一定的参数, 即可获得本体映射的结果。
本研究以UMLS包含的领域本体MedDRA与ICD为研究对象, 进行基于NCBO的本体映射实现。MedDRA对生物制药开发、临床试验、不良事件报告中的数据进行编码, 其主题包括体征、症状、疾病、诊断、治疗的适应症等。ICD为国际疾病分类。MedDRA与ICD的映射是构建相关知识库和提供临床决策的重要前提, 例如在许多电子病历系统中, 疾病利用ICD进行编码, 而其他系统中的药物不良反应使用MedDRA编码, 当需要筛选出电子病历中的哪些疾病是由药物的不良反应引起时, 即需要MedDRA与ICD映射的知识, 并结合其他药物本体才能完成知识推理的过程。
笔者使用的MedDRA版本为12.0, ICD的版本为ICD9CM, 研究MedDRA 12.0和ICD9CM之间的整体映射。
利用NCBO的REST服务实现对MedDRA与ICD9CM的映射。REST方法的具体步骤如下:
(1) 获取APIKey
首先在http://bioportal.bioontology.org/login网页注册, 在“Account”处获得“APIKey”。
(2) 获取两个本体的本体代码(Ontology ID)
要获得两个本体之间的整体映射, 必须先获得参数sourceontology={source ontology id}与targetontology={target ontology id}, 即来源本体和目标本体的代码。首先利用URI“http://rest.bioontology.org/bioportal/ontologies?apikey=YourAPIKey”得到BioPortal中所有本体的相关信息, 然后查找“MedDRA”和“ICD9CM”, 获取各自的本体代码信息。实例中, MedDRA的本体代码为:1422, ICD9CM的本体代码为:1101。
(3) 获得两个本体之间的映射结果
获得本体代码后, 即可以利用URI获得两个本体之间的映射结果。URI为“http://rest.bioontology.org/bioportal/virtual/mappings/ontologies?sourceontology=1422&targetontology=1101&pagesize=10000&pagenum=1&apikey=YourAPIKey”。参数pagesize设为10 000;当参数pagenum =1时, 返回的结果是第1页, 当pagenum=2时, 返回的结果是第2页, 依此类推。
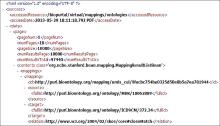
MedDRA与ICD9CM两个本体的映射结果以XML显示, 如图1所示:
此部分研究是对上述基于NCBO的本体映射研究的一个补充和拓展, 旨在改善对NCBO本体映射结果的可用性。REST的结果资源表示形式通常是XML。XML提供了适合表示半结构化数据的松散的树型结构, 已经成为当今网络数据传输、交换和存储的主要格式之一。为了获得结构化数据以更好地利用XML结果, 尤其是语义计算, 必须对XML进行解析, 以得到结构化数据。
XML文件的解析是将XML文件中的半结构化结果提取并转换成结构化格式的过程。当需要的映射仅限于不同本体之间的某些术语映射时, 返回的结果是比较易于解析的, 可以利用易操作性较强、编程方法相对简单的DOM方法。但当结果为两个本体之间的整体映射时, 可达到万条以上, DOM 方法并不适合, 因为DOM方法需要一次性读入整个XML 文档, 然后在内存中创建DOM 树, 生成DOM 树上的每个Node 对象[ 6], 程序运行缓慢, 效率低下。
因此对上述基于NCBO的XML映射结果的解析方法采用SAX方法, 尽管SAX方法在易操作性上不如DOM简便, 程序编写具有一定难度, 但其属于轻量级、由一组接口和类构成的方法, 是一种基于事件和回调模式的处理方式。针对本研究中的大量结果进行XML解析时, SAX方法具有速度快, 适用性强的优点。
XML解析后的部分结果如表3所示:
| 表3 XML解析后的部分结果 |
MedDRA与ICD9CM映射的概念通过相应的代码相互联系在一起, 为语义互联提供了知识来源。与图1中的半结构化结果相比, 表3中的数据是结构化的, 两个本体中的映射代码一一对应, 为本体互联、语义互操作中的语义信息处理, 尤其是语义计算及数据挖掘提供了数据基础。
20世纪60年代“反应停”(Thalidomide) 事件之后, 许多国家引入了药物警戒(Pharmacovigilance) 系统对上市药品进行监测。例如美国食品药品监督管理局(Food and Drug Administration, FDA) 的不良反应报告系统(FDA Adverse Event Reporting System, FAERS) 主要用于发现那些在临床试验阶段由于出现频次低而没有被识别出的罕见严重不良事件。如果在FAERS中发现潜在的安全问题, FDA将进行药物流行病学研究以进一步评价该不良事件, 确定药物与不良事件之间的因果关系。基于对药物不良事件的安全评价, FDA可能采取一系列的法规调整以提高产品安全及保障公众健康, 如更新药品说明信息、限制使用药品、向公众介绍新的安全相关信息, 或在少数情况下, 从市场上撤销该药品[ 7]。对自发病例报告的分析, 包括对FAERS的分析, 是发现上市药物未知的安全性问题的最重要方法之一[ 8]。
药物不良反应报告-数据挖掘系统(AERS-DM) 是笔者基于FAERS而获得的一个规范化数据挖掘系统[ 9], 该系统包括2004年到2011年美国FDA网站发布的药物不良反应报告;MEDI是美国Vanderbilt大学提供的一个药物适应症资源[ 10], 包括13 304对药物和适应症, 以及2 136个成分药物。AERS-DM中药物不良反应使用MedDRA编码, MEDI中药物适应症利用ICD编码。FAERS属于自发性病历报告, FAERS中既包括专业人士, 也包括患者本人提交的报告, 因此对药物不良反应的报告可能存在差错, 即患者自己提交的不良反应报告可能是药物的适应症(药物治疗的疾病) , 这样对药物不良反应的数据挖掘构成障碍, 因此需要利用MEDI提供的药物适应症进行数据清洗。数据处理如下:
(1) 对FAERS中2004年至2011年的数据进行去冗余和数据规范处理, 得到AERS-DM, AERS-DM包括2 337 414条报告, 3 422个成分药物和2 654 400对药物和不良反应;
(2) 基于NCBO进行本体映射, 将AERS-DM中药物不良反应使用的MedDRA代码映射到MEDI使用的ICD代码, 再对映射结果进行Java解析;
(3) 用Perl语言编程, 以上述解析结果为参照依据。利用MEDI中的药物适应症, 对AERS-DM中的药物不良反应报告数据进行分析, 从而在AERS-DM中筛除掉可能报告的药物适应症。实验结果显示, 删除含有可能药物适应症的报告187 938条, 包括2 663对药物和不良反应, 以及998个成分药物。
经过本体映射和解析处理后获得的数据, 作为结构化的数据格式, 是删除掉含有可能药物适应症的报告的重要知识来源, 处理后的AERS-DM将提供更高质量的药物不良反应报告数据, 从而为进行更加准确的数据挖掘提供基础。
本文在对NCBO项目引进介绍的基础上, 利用实例应用进一步展示了NCBO本体映射项目的应用价值。以MedDRA与ICD9CM为研究对象, 利用NCBO项目实现了本体映射, 并通过Java编程实现了对XML结果的解析, 将半结构化数据转换为结构化数据, 为实现语义互操作中的语义信息处理, 尤其是语义计算及数据挖掘提供了数据转换的参考方法和数据基础。本文研究也揭示了NCBO本体映射REST服务的特点, 即以机器可读的形式, 提供对生物医学领域本体映射的新型技术与工具。在未来的研究中, 将以本文所得数据为基础, 进行基于语义推理的数据挖掘。本研究所得的所有数据可向作者索取。
NCBO不仅实现了NCBO、UMLS与OBO三个资源内部基于顶层本体统控的本体映射, 也实现了三个资源间的异构本体之间的映射。可以说, NCBO在某种程度上克服了目前本体建设中存在的领域限制、数据异构和互操作不足等限制, 为领域本体融合提供了一种新颖的构建模式, 不仅对于生物医学领域数据的融合、挖掘具有重要作用, 对其他学科的本体映射与语义互联研究也起到了一种引导和促进的作用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|