作者贡献声明:
王颖: 文献调研, 论文撰写;
吴振新: 提出研究思路和论文框架;
谢靖: 文献调研。
【目的】对典型科技文献语义检索系统进行调研和总结。【文献范围】利用Web of Knowledge和Google Scholar检索Semantic Search相关文献以及语义检索系统的参考文献和研究报告。【方法】根据文本语义处理程度, 将这些系统归纳为语义查询扩展的检索系统、以概念或实体为中心的检索系统、以关系为中心的检索系统和面向知识发现的检索系统。【结果】提出科技文献语义检索系统的基本框架, 总结科技文献语义检索系统功能特点。【局限】缺少对语义检索系统的性能评测。【结论】为构建面向科技文献的语义检索系统提供良好借鉴。
[Objective] To investigate and summarize the typical semantic retrieval system for scientific literature.[Coverage] Use literatures related to semantic search retrieved by Web of Knowledge or Google Scholar, references and research reports of semantic retrieval systems.[Methods] This paper classifies current systems into four categories according to the degree of semantic processing, semantic query expansion retrieval system, concepts or entities centered retrieval system, relation-centered retrieval system, and retrieval system for knowledge discovery.[Results] The authors propose a basic framework of semantic retrieval systems for scientific literature, and summarize the features of semantic retrieval systems for scientific literature.[Limitations] Lack of performance evaluation of semantic retrieval system.[Conclusions] It provides a good guide for developing a semantic retrieval system for the scientific literature.
语义检索是信息检索的发展趋势, 早在20世纪80年代, 语义检索的思想就已经出现, 并且信息检索领域已经开展了相关研究工作。企业级的语义搜索引擎近几年已经开始应用, 例如Kosmix (http://www.kosmix.com)、Cuil (http://www.cuil.pt)、Hakia (http://www.hakia.com)和Powerset(http://www.powerset.com)等, 特别是Wolfram Alpha (http://www.wolframalpha.com)、Google Knowledge Graph (http://www.google.com)等让搜索变得更智慧。百度框计算(http://www.baidu.com)、搜狗知立方(http://www.sogou.com)代表了国内搜索引擎在该领域的成功实践。在文献信息检索领域, GoPubMed(http://www.gopubmed.com)作为语义检索系统的典型代表, 做出了开创性的工作, 一些面向科技文献的语义检索系统不断出现。
传统基于关键词的检索系统具有一定的局限性, 如无法解决词汇的模糊性问题, 分散在多个文档中的相关信息不容易被发现等。语义检索基于含义而不是通过关键词匹配寻找用户查询的答案, 用以实现实体检索、概念检索、分类检索、关系查询等知识检索方式来满足用户的多种信息需求, 使得搜索智能化, 根据用户的意图给出用户想要的结果。目前, 语义检索主要有两个方向: 语义网资源的检索和对于传统检索系统的语义扩展。面向科技文献的语义检索研究主要偏向于后者, 利用语义技术改进传统文献检索系统, 利用叙词表、主题词表、本体等知识组织体系实现语义丰富化, 采用语义标注、自动抽取、关系发现的文本挖掘技术从非结构化的文本中发现细粒度的数据, 使得检索系统更智能化。本文根据文本语义处理程度对科技文献语义检索系统进行分类, 提出科技文献语义检索系统的基本框架, 并探讨科技文献语义检索系统的功能特性。
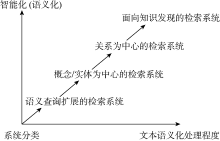
根据系统的智能化、语义化程度, 将现有科技文献语义检索系统分为: 语义查询扩展的检索系统、以概念或实体为中心的检索系统、以关系为中心的检索系统、面向知识发现的检索系统4种类型。这4类检索系统对科技文献的文本语义化处理程度不同, 检索系统的智能化和语义化程度也不同, 如图1所示:
语义查询扩展的检索系统在传统关键词检索基础上, 对检索词进行处理, 利用受控词表和本体对检索词进行扩展。PubMed[1]支持基于MeSH的查询扩展, 也有利用UMLS的同义词对PubMed查询进行扩展[2], QuExT[3]执行面向概念的查询扩展, 检索结果根据用户预先分配给概念类别的不同权重进行排序。GO2PUB[4]利用基因本体中术语之间的语义继承对PubMed查询进行语义扩展, 基因名称、符号和同义词都作为额外的关键词提交给查询处理器。
以概念或实体为中心的检索系统利用本体、主题词表、叙词表等对科技文献进行语义标注, 识别文献中的知识, 检索过程通过匹配用户查询和语义标注结果执行, 这使得检索系统能够利用标注信息查询到更精确的结果。GoPubMed[5]是这类系统中最典型的, 它利用Gene本体和MeSH标引PubMed文献, 并用于检索结果的结构化展示, 可以让用户看到与查询相关的主要的生物医学概念。相比PubMed, GoPubMed可以更快地找到相关的检索结果。NextBio文献检索系统[6]利用基于本体的语义工具和创新界面, 对Science Direct内容和PubMed、临床实验、生物医学新闻等授权开放使用的研究数据进行文本挖掘, 并通过自然语言处理技术实现命名实体识别和消歧, 从而提高检索性能。Kleio系统[7]对文本的语义概念(如genes、protein和其他生物医学术语)进行标注, 提供对于MEDLINE的文本和元数据相结合的检索, 利用标注的命名实体类型对检索结果进行分面, 从而实现检索结果的过滤。
以关系为中心的检索系统通过文本挖掘技术从科技文献中发现概念或实体之间的关系, 能够提供基于关系的检索服务。Quertle[8]是一个关系驱动的生物医学文献检索工具, 使用基于语义的自然语言处理方法从生物医学文献集中抽取主谓宾关系, 发现生物医学实体(如疾病、基因、药物)之间的一般或特殊关系。用“ 咖啡因偏头痛” 作为搜索词, Quertle会发现两个检索词之间的关系如“ 咖啡因治疗偏头痛” , 而不是通常搜索PubMed所返回的同时包含“ 咖啡因” 和“ 偏头痛” 两个检索词的记录。CoPub[9]是以共现关系为中心的检索工具, 利用文本挖掘技术检测PubMed摘要中共现的生物医学概念, 如基因本体中的人类/鼠基因、生物过程、分子功能、细胞组成以及病理、疾病、药物和途径等。在CoPub系统中检索某个生物医学概念, 可以获得与其共现的其他生物医学概念以及共同出现的文摘。PolySearch[10]抽取人类疾病、基因、突变、药物和代谢物之间的关系, 利用各种文本挖掘和信息检索技术对内容摘要、段落或句子进行识别和排序, 支持面向十几个不同类型的文本、科学文摘或生物信息学数据库的50多种查询类型, 例如检索“ 与乳腺癌有关的基因” 。
面向知识发现的检索系统通过发现隐含的关系和知识, 从而为用户提供更深层次的语义检索服务。CoPub 5.0[11]在CoPub共现关系挖掘的基础上开发了称为CoPub Discovery的新技术, 从文献中挖掘间接关系, 用于研究疾病背后的机理、连接基因和途径, 发现现有药物的新型应用等。CoPub 5.0提供了三种分析模式, “ term search” 模式为一个术语检索文摘和术语关系, “ pair search” 模式分析术语对之间的已知关系或新关系, “ set terms” 模式用以给出多个术语之间的关系。FACTA++[12]从MEDLINE文摘中发现并可视化如基因、疾病、化合物等生物医学概念之间的间接关联, 利用机器学习模型发现文本中的生物分子事件, 利用概念之间的共现关系统计出信息挖掘隐藏的关联。EvidenceFinder[13]实现对PMC全文数据从化合物基因、蛋白质、疾病等生物医学实体到如磷酸化、绑定、激活等生物相关性事实的多层次文本标注。EvidenceFinder将标注事实转化为一系列的问题, 作为文献检索的推荐, 帮助用户找到问题答案对应的文章。例如, 输入检索词“ 粘蛋白” , 系统自动给出一系列相关问题, 如“ 降低肠道粘蛋白的是什么?” 、“ 什么产生粘蛋白?” 等。
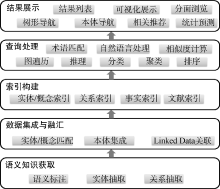
根据对典型科技文献语义检索系统的分析, 提出系统基本框架, 分为语义知识获取、数据集成与融汇、语义索引构建、查询处理、结果展示5个主要的系统功能, 如图2所示。实现科技文献的语义丰富化, 基于领域叙词表或本体, 利用语义标注、实体抽取、关系抽取等技术从科技文献文本信息中获取语义知识。以这些语义知识为基础, 借助实体或概念匹配、本体集成、Linked Data之间的关联实现潜在语义知识、科技文献以及外部资源的数据集成与融汇, 支持细粒度的语义检索以及相关知识的扩展检索。在文献元数据索引的基础上, 构建实体、概念、关系、文本事实依据的索引, 支撑基于语义的检索功能。在查询处理方面, 采用术语匹配、自然语言处理、相似度计算、知识库图遍历、本体推理等技术手段理解用户的搜索意图, 通过基于语义知识的分类、聚类、排序等对检索结果进行重新优化计算。通过结果列表、可视化展示、分面浏览、树形导航、本体导航等方式将检索结果展示给用户, 同时提供基于语义知识的相关推荐和统计预测等功能。
语义信息的引入影响了科技文献检索系统从数据处理、索引构建、查询处理到结果管理的各个方面, 使得检索系统具有一些新的特性。
在传统文献标引的基础上, 一些文献检索系统已经进行了深层的语义丰富化处理, 并且在此基础上提供更准确的检索服务。例如, ProQuest①(①http://search.proquest.com/)在文本标引基础上将蕴含在学术出版物中的表格、地图、照片和其他图形中的数据、变量以及其他内容进行深度的标引, 平均使用8个术语描述一个图像。Wiley的Smart Article技术[14]针对化学期刊新增了化合物索引, 提供对于内容的深层检索, 此外对文献中的化学术语进行标注, 使用不同颜色对不同类型的化学术语进行高亮显示, 以方便用户阅读。在医学文献检索领域, PubMed使用MeSH主题词表进行文献标引, 随着文本挖掘技术的成熟, 一些工具和系统在PubMed基础上对科技文献进行了更为深入的语义丰富化处理[15]。例如, EBIMed[16]从文献中抽取蛋白质、基因本体标注、药物和物种, 基于共现分析识别抽取概念之间的关系。PubTator工具[17]支持对PubMed检索结果的标注, 识别的生物医学实体包括基因、化学物质、疾病、变异、物种等。
科技文献的数据集成已转变为以实体或概念为中心的数据集成和融合, 实现不同应用系统之间的语义互操作, 促进更广泛的共享与应用。AGRIS国际农业科学和技术信息系统利用OKKAM实体名称系统框架[18]创建关联数据模型, 将书目数据库转换为关联数据服务[19]。一方面, 使用AGROVOC叙词表与其他叙词表映射, 另一方面将书目记录与外部资源建立连接, 如DBPedia、WordBank、Google Custom Search API、Nature OpenSearch等。在AGRIS检索结果的详细页面中, 除书目信息外, 还提供相关外部资源的结果揭示, 借助文献标引使用的AGROVOC词汇、书目关联数据等实现以实体或概念为中心的知识页面之间的融汇。Elsevier提出Smart Content的概念[20], 组织医学专家在UMLS基础上构建EMMeT医学词汇分类体系, 将Elsevier的临床医学期刊、论文、书目章节、表格、图像等数据映射到合适的医学术语上, 从而加强对Content的理解, 使其提升到实体、概念和关系的知识层面上, 以便各类应用程序更好地理解和处理内容上的内涵信息。
为实现对文本分析结果的检索, 语义检索系统构建了文本中概念、实体、关系、事实与文献之间的索引。例如, Kleio系统应用Lucene对识别出来的蛋白质、基因、代谢物和医学术语构建索引, 即对与文本相关的概念构建索引, 而不是个体或规范词形式, 这意味着系统可以检索与某个指定概念相关的文档, 无论概念的表现形式是它的拼写变体还是缩写形式[7]。EvidenceFinder系统[13]借助基因、蛋白质、药物、疾病和代谢物的词表以及表示生物医学过程和关系的词典, 对Europe PMC仓储全文数据进行语法分析和文本挖掘, 将所有可能包含相关事实的句子构建索引。NLMplus[21]使用Solr对语义层进行索引, 支撑检索服务。而Quertle[8]建立语义关系索引、关键词索引和辅助索引三种索引, 用于查找用户输入的检索词和提问, 并返回检索结果。
由于一个搜索请求可能代表多重含义, 对用户输入的检索词进行语义分析是语义检索系统的首要任务。通常, 语义检索系统从用户输入字符开始提供自动完成功能, 对用户输入的检索词和语句进行识别和分析, 给出相关的查询建议, 通过理解用户查询意图和搜索空间的含义改进检索质量。
(1) 基于受控词表和本体的自动完成功能
目前, 搜索引擎大多数都具有自动完成功能, 利用预存的术语自动将用户的检索词对应到可能匹配术语上并提示给用户, 简化用户输入操作。文献检索系统通常利用受控词表和本体实现自动完成功能, GoPubMed[5]将输入的术语匹配MeSH和Gene本体术语; Semedico[22]将查询建议放在分类树中允许用户选择一个广义术语作为检索词, 在括号中列出其同义词; NextBio[6]可以列出匹配的基因、化合物、SNPs、疾病、组织、生物学团体和作者等; Elsevier的ClinicalKey医学信息平台①(①https://www.clinicalkey.com/.)在用户输入检索词后提供检索建议, 如相关医学主题、内容来源和作者等。
(2) 查询分析
检索系统在执行查询前, 采用语言学方法将用户输入的检索词映射到受控词表或本体的概念、实体上, 将关键词检索转化为概念或实体的检索。利用受控词表的同义、广义、窄义等术语以及基于本体上下位关系实现查询的逻辑推理, 用于解释用户的查询, 并给出查询建议。Kleio系统将摘要中命名实体进一步分类, 结合语义分类信息执行查询, 可以降低搜索空间, 提高检索效率[7]。一些文献检索系统允许用户使用自然语言进行提问, 如Quertle、EvidenceFinder等, 在执行查询处理前, 需要对查询语句进行预处理, 利用自然语言处理技术将查询语句进行重构。NLMplus[21]使用叙词表和本体对PubMed Review进行语义标引, 利用构建的知识库对查询进行分析和解析, 以检索到更精确的结果。iPubMed[23]提供一个交互式检索界面, 当用户在搜索框中输入几个字符时, 系统将立即显示任何包含这些字符的引用, 便于缩小搜索目标, 此外该系统还允许小的拼写错误。ClinicalKey通过EMMeT建立关系的语义框架, 促进内容发现, 使得被传统关键词检索忽略的潜在关联能够被揭示出来, 并且保证了ClinicalKey能够为用户的检索请求提供具体并且有针对性的答案, 比如查找“ myocardial infarction” , ClinicalKey智能检索可以识别其缩略词、同义词、相关外科手术和治疗药物, 并且知道这是一种与高胆固醇相关的心血管疾病[20]。
在传统文献检索系统的基础上, 语义检索系统对于查询结果的呈现方式更加多样, 表达的信息也更加丰富, 基于本体的结果精炼、知识导航等为用户带来了新的检索体验。
(1) 查询结果呈现方式
语义检索系统为用户提供了最直接的结果呈现方式, 如检索的目标概念(实体)、关系、事实、回答等信息。GoPubMed[5]在文献结果列表中只显示文摘中与检索目标相关的句子, 反映检索词的事实, 而不是全部摘要信息。Quertle[8]同样显示文摘中相关的事实信息, 并对检索目标进行高亮显示。FACTA++[12]将与查询目标相关的概念通过不同分类列表的方式显示, 并可以按照相关的频次排序。CoPub[9]返回查询术语的详细信息、共现术语的分类和文摘数量。EvidenceFinder[13]在文献检索列表中直接给出查询问题的答案并高亮显示。
(2) 概念/实体层级结构分类与导航
GoPubMed[5]通过本体的层级结构对查询结果进行聚集, 实现了大规模结果的快速导航, 用户可以快速获取相关的生物医学概念, 同时可以在检索中发现新的检索目标或过滤检索条件, 使得检索更有深度和广度。NextBio[6]将从摘要和正文中抽取的生物医学术语, 以Tag云的方式显示, 并提供这些术语的分类, 可以利用它们进一步过滤和优化查询结果。Kleio[7]将检索结果根据文献标注命名实体的语义分类进行组织, 并列出最高关联频率的概念, 方便用户浏览和过滤检索结果。ClinicalKey允许用户根据有临床意义的子分类筛选检索结果, 比如内容类型、专科、疾病名称、身体部位等[20]。
(3) 文本挖掘结果显示与相关知识导航
在结果页面或文献详细页面对语义标注结果进行呈现, 并提供相关知识的简介、链接与导航, 例如GoPubMed[5]在标注概念下方用虚线标记, 点击后可实现对标注概念的重新检索和二次检索, 以及直接给出标注概念的详细信息、Wikipedia链接。EvidenceFinder系统[13]在文献详细页面将识别的生物实体统计情况以图形化的方式显示, 并根据不同的类型分别列出, 点击标注实体可以直接链接到UniProtKB①(①http://www.uniprot.org/uniprot/.)的相关检索界面, 查看相关信息。ClinicalKey平台在检索结果页面提供文献摘要的预览窗口, 同时对语义标注的结果进行展示, 并且提供2 000多个疾病主题页, 可以快速访问疾病的流行病学、风险因素、临床表现、治疗等方面的信息, 以及与特定专科相关的答案和药物链接[20]。
(4) 基于概念/实体的文献统计分析
通过对文献的文本挖掘, 语义检索系统可以实现基于概念/实体而不是关键词等元数据信息的文献统计分析功能。例如, 在GoPubMed[5]平台上点击左侧导航的概念或文本标注概念都可以看到该概念相关文献的时间轴, 不仅可以展示相关文献的演化过程, 也可以预测其发展趋势。
科技文献语义检索系统相比传统检索系统, 其优势在于能够处理语义信息, 从非结构化文本中发现潜在知识, 实现知识检索, 满足用户更高的检索需求。通过研究和分析现有科技文献语义检索系统可以发现系统的语义化程度依赖于对文献的语义挖掘深度, 借助现有的文本挖掘、自然语言处理、语义网等技术以及受控词表和本体, 在很大程度上实现了对指定信息的挖掘和发现, 然而由于受控词表和本体的领域局限性和覆盖率问题, 科技文献语义检索系统的研究主要集中在生物医学领域, 而在科技文献检索领域实现通用的语义检索仍然困难重重。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|