作者贡献声明:
钱庆: 提出Hadoop/HBase云存储的研究思路;
方安: 准备研究环境, 设计研究思路;
范云满, 洪娜: 实施实验, 收集数据, 起草论文;
洪娜: 论文修改及最终版本修订。
【目的】探索在导入、保存、检索、批量导出生物医学大数据方面的新思路和新方法, 积累第一手经验。【方法】分析生物医学大数据的特点, 从理论方面和数据查询对比实验两个方面, 对比分析以Oracle为代表的传统关系数据库和以HBase为代表的NoSQL数据库在解决大数据问题时各自采用的技术以及各自的优势与不足。以一个药物基因组数据存储系统为例, 进行云存储实践和初步的对比实验。【结果】HBase在处理大量数据的实际应用中, 比Oracle更具优势。【局限】没有对药物基因组学数据进行深入挖掘分析, 同时需要对Hadoop/HBase做深入的技术优化。【结论】HBase在本文实验的应用场景中能够满足生物医学大数据存储的要求。
[Objective] To explore the new idea and method, accumulate first-hand experience from the aspects of importing, storaging, retrievaling and bulk exporting the large-scale biomedical data.[Methods] Analyze the characteristics of the large-scale biomedical data, and compare the technologies, the advantages and disadvantages for solving the big data problem of the traditional relational databases (the representative Oracle) and the NoSQL database (the representative HBase), from the aspects of theoretic and test results. Take a drug database of genomic data storage systems as an example, and make a test for the performances of Oracle and HBase.[Results] HBase in practical application has a large advantage over Oracle when process large data.[Limitations] Lacking the deep mining and analysing to the pharmacogenomics data, the future research needs an in-depth technical optimization for Hadoop/HBase.[Conclusions] In this experiment, HBase can meet storage requirements for the large-scale biomedical data.
随着现代信息处理技术的发展, 大数据时代已经到来, 其中生物医学领域面临的大数据问题是具有代表性的一个分支。生物医学数据具有以下特点:
(1) 生物医学领域的公开数据集非常多。生物医学领域关乎整个人类乃至整个生态系统的生存与发展, 各个国家与机构对于数据管理大多采用完全共享(PubMed[1])、有责任的共享(UMLS[2])等机制。
(2) 生物医学领域数据的种类非常多, 如基因数据、蛋白质数据、疾病数据、核酸数据等。
(3) 生物医学数据的数据量巨大。如UniProt[3]数据的蛋白质知识库包含Swiss-Prot和TrEMBL两个数据集, 其中Swiss-Prot是经过人工标注和审核过的数据, 包括546 790条; TrEMBL是经过机器标注的, 没有经过审核的数据, 截至2014年10月30日已有86 536 393条数据(http://web.expasy.org/docs/relnotes/relstat.html)。
(4) 生物医学数据增长迅速。如UniProt中的TrEMBL数据, 其数据量从2008年开始快速增长, 如图1所示, 从400多万条增长到8 600多万条, 年均增长率为66.8%。
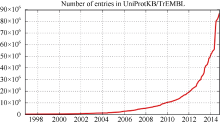 | 图1 UniProt中TrEMBL数据的数据量曲线(数据来源: http://web.expasy.org/docs/relnotes/relstat.html, 采集时间: 2014年10月30日) |
虽然已有研究人员对NoSQL数据库的应用做了研究[4, 5], 分析总结各种NoSQL数据库在测试时性能方面的表现[6, 7, 8, 9], 并基于这些数据库做了相应的实践探索[10, 11, 12, 13]。但是这些探索存在一些不足, 研究往往限定在一种NoSQL数据库的应用实践, 没有对NoSQL数据库和SQL数据库的性能进行测试对比。本文从在不同数据量级下的单条数据查询、多条数据查询及不同数据量级下不同字段个数、不同数目的批量数据加载多个角度进行深入对比分析, 能够弥补前人研究在对比分析方面的不足, 从而为数据仓库建设提供理论依据, 能够为进一步建设生物医学数据云存储提供支撑, 具有一定的实践意义。本文仅对药物基因组学数据做了初步的关联, 为大数据量的存储提供前期的探索与技术的储备。
传统关系数据库(RDBMS)是将数据按照二维表的形式进行按行存储, 每个二维表代表数据的一个实体, 实体与实体之间的关系采用外键的形式进行关联。数据的操作采用结构化查询语言(Structed Query Languag, SQL)。Oracle数据库是甲骨文公司的一款最具有代表性的关系数据库产品, 具有优良的性能。通过索引、二级索引、外键以及纵向分区、横向分区和分片等特性, 提供对关系数据查询的快速响应、数据关系组织的存储支持。
但是以Oracle为代表的关系数据库在面临大数据挑战时, 逐渐暴露出自身问题:
(1) 分区操作导致成本提高和系统复杂度上升。
(2) 关系数据库是按行存储, 即使是查询一个表的一列数据, 也需要读取整个表所有列的数据, 加大了数据库I/O的负担, 从而操作的时间相应变长。
(3) 关系数据库在对数据存储方案(Schema)修改时, 容易导致较大的空间浪费。
(4) 即使采用Oracle RAC机制, 对于数据库中数据的操作始终也是在一个共享的文件系统中, 所以这种方案最大的问题仍然是磁盘的I/O问题没有得到彻底解决。
(5) Oracle是一款商业化产品, 其License按照CPU数量收费, 购买License是一个巨大的成本。
(6) 一般运行Oracle数据库的服务器为了达到较高的性能和可靠性, 会采用小型机甚至大型机, 因此硬件成本非常高。
HBase是和Hadoop紧密相关的一种NoSQL数据库[14]。它利用Hadoop的分布式文件存储系统存储数据。HBase具有以下特点:
(1) 数据按列存储[15], 在查询数据时只需要查询需要的列, 不需要读取所有列, 从而提高检索速度。
(2) 基于Hadoop的平台可以充分利用分布式计算与存储的优势, 将读取数据的I/O瓶颈分解到各个DataNode加以解决。
(3) HBase在存储时采用内存和物理存储相结合的方式, 提高了读取速度。
(4) 由于HBase运行在Hadoop上, 而Hadoop可以部署在多台廉价的普通PC Server上, 节省了购置硬件的成本。
(5) HBase是完全开源的免费软件, 省去了购买License的开支。而这些是进行科研项目时需要考虑的问题。
从表1可以看出, Oracle适合保存结构化数据, 但是由于其集中化存储无法解决I/O性能问题, 所以与HBase这类支持分布式存储的NoSQL数据库相比, 批量读取的性能相对较弱。同时, Oracle是一款商业化数据库, 购买License需要花费很高的费用; 并且Oracle一般需要部署在高昂的小型服务器甚至中型服务器上, 硬件成本同样很高; Oracle虽然是成熟的商业化软件, 使用者与开发者众多, 但是这类NoSQL数据库由于是开源型数据库, 近年来发展非常迅速, 使用者与开发者增长迅猛, 导致基于两者进行各自相应的开发与维护成本不相上下。综合而言, 以HBase为代表的NoSQL数据库在综合成本上占据较大的优势。
| 表1 Oracle与HBase数据库的特征对比 |
在数据导入时间、不同数量级以及不同查询复杂程度下的查询时间、数据抽取时间等几个方面对Oracle和HBase的性能进行对比分析, 为后续数据存储中的实验做技术准备。本文将UniProt[3]发布的Swiss-Prot数据中的化合物(Compound)、基因(Gene)、疾病(Disease)数据作为测试数据, 为了满足大数据量的测试, 对每种数据复制多次, 以分别达到百万、千万、亿、十亿条记录。为了取得一个比较稳定准确的时间数据, 对于每种测试重复三次, 取其平均值作为该次测试的时间数据。
(1) 数据导入时间
数据导入时间的对比如表3所示:
| 表3 数据导入时间对比 |
其中, HBase的导入采用JDBC连接方式花费时间最长, 采用BulkLoad(批量导入)方式花费时间接近于Oracle的一半。因此, 在批量数据导入方面, HBase比Oracle更具时间优势。
(2) 数据查询时间
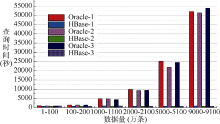
图2对比了Oracle和HBase在5种情况下的查询时间: 单表单条查询(-1)、两表联合查询单条(-2)、三个表联合查询单条(-3)、两个表联合查询多条(-4)、三个表联合查询多条(-5)。可以看出, Oracle在上述5种情况下, 对数据的查询速度影响不大, 但是HBase呈现出一些与Oracle不同的性能表现, 具体体现在: 随着每次数据量级的变化, 相应的查询时间缓慢上升; 随着查询复杂度的上升, 查询花费的时间上升的速度较快, 远远大于由于数据量级导致的查询时间的上升。这说明在上述5种情况下, Oracle在查询速度上的性能远远优于HBase, 同时Oracle对于查询单条记录还是少量的多条记录, 对于数据量级的影响, 以及对于查询复杂度的敏感程度都远远低于HBase。
(3) 数据抽取时间
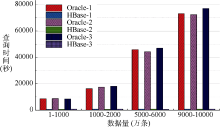
为了测试两种数据库抽取数据的性能对比, 本文从不同数量级下, 从一个一亿条数据的具有20个字段的表中抽取100万条、1 000万条的部分字段分别进行测试, 结果如图3和图4所示, 其中后缀-1、-2、-3分别表示取3个、5个、10个字段。
从图3和图4可以看出, Oracle在批量抽取数据方面性能显著不如HBase, 具体表现在: 随着抽取数据范围的变化, 抽取所占用的时间呈现快速上升的势头, 而HBase则对于数据范围在整个数据表中的区间位置不敏感; Oracle和HBase在抽取字段个数上对于查询时间的影响不是非常明显。造成上述现象的原因是由于Oracle是按行存储, 从一个表中无论抽取多少个字段, 即使抽取一两个字段, 都需要将表中相关的数据全部扫描一遍, 因此抽取不同的字段对查询时间的影响不明显; 同时, Oracle抽取不同区间位置的数据时, 需要定位到该区间, 而这也需要较长的时间, 导致在批量抽取数据时, 随着数据所在区间位置逐渐后移, 花费时间也迅速上升。可以看出, Oracle在批量抽取大规模数据时, 即使抽取较少的字段, 在抽取速度上都远远不及HBase, 而HBase作为数据仓库, 批量抽取大规模数据(在本次实验中分别为百万级、千万级)则具有更大的优势。
(4) 数据存储空间
数据存储空间的大小间接反映了数据库对于数据存储的效率, 较高的存储效率能够在存储海量数据时节省大量的存储空间, 从提高硬件和资金利用率角度而言, 具有巨大意义。为了测试Oracle和HBase的存储效率, 同时考虑实际的硬件测试环境, 本文选择一个具有20个字段1亿条数据的表, 测试Oracle和HBase存储该数据时占用硬盘空间的情况, 结果如表 4所示:
| 表4 Oracle和HBase数据存储空间对比 |
在Oracle中数据表存在两个索引, 占用数据空间15.33GB, 索引占用空间2.88GB, 总计占用空间约18.21GB; 将该数据导入到HBase中, HBase中只有一个主索引, 不能创建二级索引, 同时主索引占用的空间无法和数据占用的空间区分, 因此总计占用空间45.93GB, 但是由于HBase存储在Hadoop之上, Hadoop本身具有数据备份和冗余的功能, 本次测试的Hadoop数据保存策略是将同一份数据保存三份, 那么在HBase中每份数据占用的空间为15.31GB, 即使不计算Oracle的索引占用空间(15.33GB), HBase在存储上也略占优势。当然, Oracle为了提高数据的访问效率, 支持建立二级索引, 在一定程度上占用了更多的空间, 如果将此二级索引占用的空间去掉(2.88GB/2=1.44GB), 虽然节省了一部分空间, 但是性能会受到较大的影响。此外, 在本次实验中, 该表不是一个稀疏表, 如果是稀疏表, 则HBase在存储空间的效率方面则更具有优势。
经过对Oracle和HBase从数据导入时间、数据查询时间、数据抽取时间和数据存储空间4个方面的对比可以得出以下结论:
(1) 数据导入速度: HBase的数据导入速度远远快于Oracle。
(2) 数据查询速度: 查询少量数据时, Oracle远远超过HBase; 抽取大量数据时, HBase远远优于Oracle。
(3) 数据吞吐速度: 由于HBase是分布式存储, 将I/O分散到多个节点, 因此HBase在这一方面优势非常明显。
(4) 数据存储空间: HBase是分布式存储, 同时又具有较高的存储效率, 因此在这一方面HBase也具有优势。
总体而言, HBase适用于大数据量吞吐的数据仓库式仓储, 而Oracle非常适合于即时交易等数据处理方面。
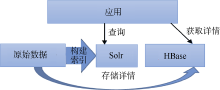
药物基因组学(Pharmacogenomics), 又称基因组药物学或基因组药理学, 主要研究遗传学在药物反应中的作用。通过将病人的基因表达或者单核苷酸的多态性与药物的疗效或毒性联系起来, 研究遗传变异对于药物反应的影响[16]。笔者前期构建了一个信息系统, 该系统尝试利用药物、基因、蛋白质等生物医学数据及其之间的关系为药物基因组学提供信息统计与分析功能。在系统实现上, 系统的存储技术架构如图5所示:
将原始数据导入到Solr①(①http://lucene.apache.org/solr/.)构建索引, 在Oracle中存储详细信息, 通过这样的存储方式, 业务应用在Solr中检索到相关的数据, 在Oracle查询相应数据的详细信息。本系统在运行时发现查询详情信息时画面载入过慢。另外, 为了在利用HBase存储上有第一手的实践经验, 本文将原始的存储技术架构改造为如图6所示的架构, 将原始数据改为利用HBase存储, 测试其在实际应用中查询速度等方面的效果。
该系统中包含的数据有基因、蛋白质、疾病、药物、化合物、文献实体和各种实体之间的相关关联信息, 如基因与疾病、疾病与药物等的关联数据、实体内部的关联数据, 如不同来源数据库的基因、蛋白质、疾病等信息。在查询某一种基因时可以查询到相关的疾病、药物、化合物、疾病等。因此在查看一条数据详情时, 需要批量加载该条数据大量的相关数据, 而这在前面的测试实验中已经显示出HBase的优势。实验系统包含的实体类别以及数据数量如表5所示:
| 表5 实验系统中包含的实体类别以及数据量 |
以基因与基因概念、基因与疾病、药物与化合物的关系为例, 测试改造前与改造后, 加载一条数据的同时并加载与之相关的大量关联数据时的时间对比。测试结果如表6所示。通过改造前后的时间对比可以反映出在批量加载大量数据时, Oracle在性能上远远不如HBase。
| 表6 改造前后加载时间对比 |
通过本次改造得到以下结论:
(1) 从技术实现的角度, HBase已经有了完整的API支持, 从而证明将基于传统的Oracle的系统改造成基于Hadoop/HBase的云存储的技术路线图是可行的。
(2) 在批量加载大量数据时, HBase在性能上远远优于Oracle, HBase更加适用于该应用场景。
在面对生物医学大数据时, 传统的以Oracle为代表的关系数据库面临着巨大挑战, 同时已经在业务应用场景中暴露出一些问题。为了探索在导入、保存、检索、批量导出生物大数据方面的新思路新方法, 笔者进行了以下的研究工作:
(1) 从理论方面对比分析了各自的性能特点, 并在随后的测试实验中进行对比, 发现传统的关系数据库在查询加载小数据量时其查询速度具有较大的优势, 但是在查询加载大量的数据时, 即使每次仅加载数据中的部分字段, 其性能远远低于HBase这类按列存储的NoSQL数据库。
(2) 由于HBase是基于Hadoop的分布式存储数据库, 其数据的吞吐速度利用多个节点的I/O得以提升, 使得在数据导入、数据抽取时间方面比Oracle有很大的提升。另外, HBase是按列存储, 有效避免了Oracle这类传统的关系数据库按行存储时由于数据稀疏带来的存储空间浪费问题。
同时本文研究的局限包括以下两个方面:
(1) 对以药物基因组数据为应用数据的实际系统中没有进行深入的挖掘分析, 未来可以借助Hadoop的数据分析平台进行数据关系的深入挖掘, 而不是限于目前的关联整合。
(2) 没有对Hadoop/HBase系统进行深入的技术优化, 这是需要尝试解决的问题, 从而为下一步的工作做好技术准备。
除去HBase数据库, 还需要探索其他的存储数据库, 使其能够更好地支持生物医学大数据, 更好地挖掘生物医学数据内部隐含的关系, 考虑采用Graph DataBase Neo4j (http://neo4j.com/)做更加深入的探讨。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|