1 引言
目前, 基于英文电子病历的相关研究已全面展开, 而基于中文电子病历的相关研究正在探索。亟需开展基于中文电子病历的标注方法研究, 以提高对健康医疗数据的分析与处理能力, 促进中国医学与健康高端智库建设。
因此, 本文以临床医疗知识发现为导向, 构建中文电子病历标注平台, 在此基础上, 开展医疗实体标注实证研究。
2 相关研究
2.1 英文电子病历
在英文临床文本标注方面, 已经开展多项针对医疗文本的标准化公开评测任务, 例如ShARe/CLEF[14]、SemEval[15]、i2b2[16]等, 为基于临床文本的数据挖掘与分析提供了基础研究数据。与此同时, 英文临床文本处理系统也得到了持续发展。基于Pipeline模块化思想[17], IBM提出非结构化信息管理架构(Unstructured Information Management Architecture, UIMA), 将临床文本处理的各个环节模块化为一组组件。UIMA包括语言识别、语言分析、字典查找、命名实体识别、模式匹配、分类模块以及用户自定义7类标注组件, 已被广泛用于各种英文临床文本处理系统, 如BioTeKS[18]、cTAKES[19]、DeepPhe[20]等。其中, cTAKES是一个大规模的开源临床文本自然语言处理系统, 具有良好的可扩展性, 旨在处理和提取语义信息, 支持异构数据的临床研究。其功能模块和语料库得到持续的丰富与完善, 在PTB[21]和GENIA[22]语料库的基础上, cTAKES还增加了来自梅奥诊所电子病历的临床标注语料。
2.2 中文电子病历
中英文自然语言处理的一个最显著区别在于中文表达与英文使用空格作为词与词之间的分隔标识不同, 分词是中文自然语言处理的首要工作。此外, 中文电子病历具有显著的半结构化文本结构和较强的模式化语言特点[9], 包含大量临床专业术语和习惯性表达。中文临床术语尚未形成统一标准, 不同医院以及不同科室的医学术语缺乏一致性约束, 电子病历结构化发展程度表现出不均衡的特点, 极大地制约了临床大数据的整合分析及深度挖掘, 从而进一步影响到临床知识发现的效率和效果。面向中文电子病历的相关研究主要集中在两个方向: 基于特征工程的病历标注; 基于深度学习的医疗信息抽取。
哈尔滨工业大学关毅教授的团队是开展中文电子病历文本标注较早的团队, 该团队在参考和借鉴2010 i2b2/VA的基础上, 构建了包括疾病、诊断、症状、检查和治疗5类医疗实体的标注体系, 并制定中文电子病历命名实体和实体关系标注规范[27], 实现了对哈尔滨医科大学附属第二医院992份电子病历的人工标注[28]。通过构建适合于中文电子病历的数据特征, 实现对医疗实体和实体关系的标注, 标注结果具有可解释性, 这对于临床应用极为重要。然而, 基于特征工程的病历标注需要有临床专业医生的参与, 特别是在标注指南制定、标注人员培训、数据标注以及标注结果的审核过程中, 需要充分发挥临床医生对医疗知识的掌握和运用能力[29]。
在全国知识图谱与语义计算大会(China Conference on Knowledge Graph and Semantic Computing, CCKS)和中国健康信息处理会议(China Health Information Processing Conference, CHIP)等的推动下, 国内已开展基于深度学习等方法的医疗实体抽取活动, 其中, CCKS 2017的任务是实现对症状和体征、检查和检验、疾病和诊断、治疗以及身体部位5类电子病历命名实体的识别[30], CCKS 2018的任务是实现对解剖部位、症状描述、独立症状、药物以及手术这5类命名实体的识别[31], CHIP 2018的任务是对医学影像学检查结果进行结构化, 识别出肿瘤原发位置、病灶大小以及转移部位[32]。这些评测任务为医疗实体识别提供了宝贵的数据资源与学习交流平台, 推动了基于中文电子病历的相关研究。基于深度学习的方法有效降低了人工标注数据的成本, 并可提高电子病历信息抽取效率, 然而其可解释性有待进一步探索。
临床实践涉及伦理道德, 临床实践结果需要具备可解释性, 因此本文基于特征工程研究中文电子病历的标注方法。为减少人工标注的时间成本, 基于少量标注病历, 通过机器学习实现病历的自动标注; 在机器自动标注的结果基础上, 通过人工辅助审核从而形成较大体量的病历标注语料; 基于该语料库, 借鉴英文电子病历文本处理中的Pipeline模块化思想, 构建从分词到词性标注再到医疗实体识别的Pipeline, 并通过实验验证本文所提病历标注方法的可行性。
3 中文电子病历标注方法
3.1 总体思路
作为临床文本资源知识化的重要环节, 病历标注需要有专业医生的指导和参与, 其质量关系到临床知识发现的效率与深度, 然而临床医生的时间和精力极其有限, 病历文本自动标注面临着人工标注语料库体量不够的挑战。为减轻人工参与的标注任务量, 本文提出一种人工辅助机器自动化的病历标注方法, 旨在基于少量标注病历, 通过综合利用自然语言处理和机器学习实现对中文电子病历的标注。总体思路如图1所示, 包括机器学习、病历标注和Pipeline三个阶段。
图1
(1) 机器学习
条件随机场(Conditional Random Field, CRF)是序列标注中常用的模型, 相比隐马尔可夫模型(Hidden Markov Model, HMM)和最大熵马尔可夫模型(Maximum Entropy Markov Model, MEMM), CRF能利用更多特征, 且更能抵抗标记偏置[33]。因此, 基于N份已得到标注的病历数据, 首先通过CRF学习得到病历标注的模型, 该模型通过评价验证有效性后, 用于数据标注阶段的M(M > N)份病历标注。
(2) 病历标注
利用机器学习阶段得到的训练模型, 采用基于单字特征的CRF算法识别病历中的医疗命名实体。由于机器标注的病历数据难以达到人工标注的精确度, 因此需要构建可视化的病历标注平台, 并基于“建模-标注-训练-测试-评价-修订(Model, Annotate, Train, Test, Evaluate, Revise, MATTER)”的循环标注方法[34], 在该平台上对机器自动标注的数据进行审校, 最终形成N + M份标注病历数据集, 从而使病历语料库的大小在原始N份的基础上扩充M份。
(3) Pipeline
通过Pipeline可将临床文本处理的各个环节模块化为一组组件, 该方式具有良好的可扩展性。基于机器学习和病历标注两个阶段得到的N+M份标注病历, 在Pipeline阶段对病历数据进行分词、词性标注和临床医疗命名实体识别, 以验证本文所提中文电子病历标注方法的可行性, 从而为基于临床文本的深度挖掘和知识发现提供标准化和结构化信息。对于Pipeline阶段, 由于前一个过程的输出作为后一个过程的输入, 因此, 分词和词性标注的质量直接影响医疗实体识别的效果。
3.2 标注平台
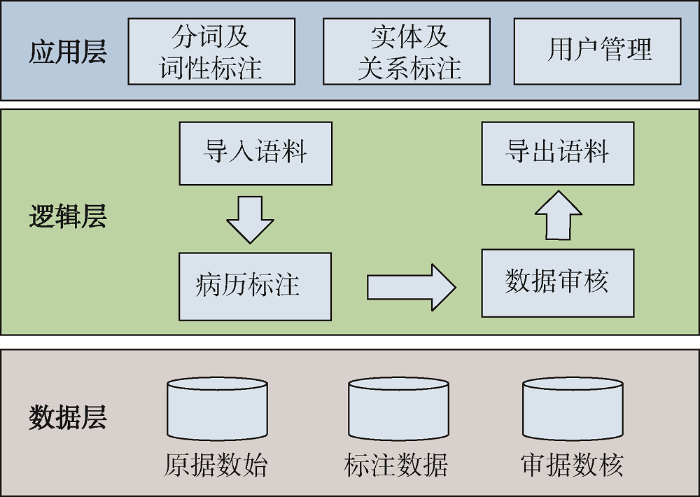
(1) 平台架构
图2
①数据层
病历标注的对象是原始电子病历数据, 通过机器学习实现自动化标注, 该标注数据需要通过专业医生和计算语言学家的人工审校之后形成规范的病历标注语料, 因此数据层包括原始数据集、标注数据集以及审核数据集。
②逻辑层
从逻辑层面, 平台首先需要为原始病历语料数据的导入提供安全接口, 支持对导入病历的自动化处理并反馈自动标注结果, 针对机器自动标注结果需提供可视化的人机交互审校界面, 为保证标注结果的可用性, 平台应支持以xml、txt、xls等多种格式导出标注病历语料。病历数据的导入和导出一般采用批量方式。
③应用层
根据研究思路, 针对中文电子病历数据, 标注平台需提供分词、词性标注以及医疗实体标注等服务, 且根据标注的各个不同阶段, 平台用户可以划分为标准组、标注组、审核组以及算法组, 平台需支持对不同类型用户的管理。
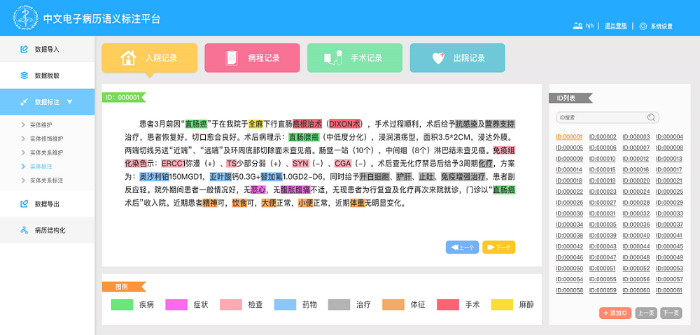
(2) 平台功能
基于上述平台架构设计实现中文电子病历标注平台, 实现效果如图3所示, 图中样例数据来源于CCKS评测语料。
图3
①数据导入: 平台以临床知识发现为导向, 支持对病历数据的导入功能, 主要包括入院记录、病程记录、手术记录和出院记录。
②数据脱敏: 由于电子病历数据包含患者和医生的隐私信息, 因此, 数据处理的首要工作是识别数据中的敏感信息并进行数据脱敏。
③数据标注: 电子病历中包含各种医疗实体, 不同实体可以对应不同的修饰类型, 实体之间可以对应多个关系, 因此, 数据标注功能包括实体维护、实体修饰维护、实体关系维护、实体标注以及实体关系标注。
④数据导出: 平台支持对标注后的结果进行导出, 通过对非结构化的文本数据进行标注, 为临床大数据的深度分析与挖掘提供规范化和结构化数据。
3.3 标注算法
(1) BEMS序列标注
采用单字特征训练临床命名实体识别模型, 将位置编码与实体编码相结合, 对医疗实体进行编码。字符在实体中位置的编码采用BEMS序列标注, 其中, B代表Begin, 即词首; E代表End, 即词尾; M代表Middle, 即词中; S代表Single, 即单字成词。例如, “伤及右髋部”可被编码如下。
其中, BODY代表身体部位, O代表非医疗实体。
(2) 分词工具
目前开源的分词工具均针对通用领域, 没有医疗领域的专有分词工具, 所以在临床文本处理过程中容易出现分词结果与实体边界发现冲突的现象。例如, 对“心肺腹未见异常”进行分词, jieba分词①(①https://github.com/fxsjy/jieba.)的结果如下。
心肺 腹 未见异常
而其命名实体标注的结果如下。
心: S-BODY 肺: S-BODY 腹: S-BODY
可以看出, 在分词的结果中, “心肺”为一个词; 而在命名实体标注中, “心”与“肺”为两个独立实体, 从而造成边界冲突。本研究比较了jieba分词和HanLP分词②(②https://github.com/hankcs/HanLP.)效果, 以CCKS 2017标注语料为例, 通过jieba分词的冲突数量达1 765个, 利用HanLP分词的冲突数量为751个。
因此, 本研究首先利用HanLP对语料进行粗分, 然后在标注平台中对分词的初步结果进行校准, 并通过CRF算法对校准之后的语料进行训练, 利用Java的SpringBoot封装Web API分词接口, 从而用于临床命名实体识别。
(3) CRF特征模板
图4
图5
Unigram生成CRF的状态特征函数, Bigram生成CRF的转移特征函数。基于词的CRF特征模板加入的特征包括前第二个词、前一个词、当前词、后一个词、后第二个词、前第二个词的词性、前一个词的词性、当前词词性、后一个词的词性、后第二个词的词性、前第二个词和前一个词的词性、前一个词和当前词的词性、当前词和后一个词的词性、后第两个词和后第三个词的词性。
4 中文电子病历标注实证——以CCKS语料为例
4.1 语料数据
采用CCKS 2017 “电子病历命名实体识别(Clinical Named Entity Recognition, CNER)”评测任务数据集。该数据集包括300份人工标注病历和400份质量较好的未标注病历, 且每份病历都包含病史特点、出院情况、一般项目和诊疗经过4个文本域。标注的目标是识别出电子病历文本中的症状和体征、检查和检验、治疗、疾病和诊断、身体部位5类实体。
4.2 标注流程
由于用于评测任务的测试集未公开, 因此, 首先基于原始已标注的300份电子病历, 通过CRF学习得到病历标注的模型, 然后利用该模型对评测数据集中400份质量较好的未标注语料进行标注。根据3.1节的标注思路, 具体步骤如下。
(1) 基于单字特征得到模型预标注数据, 在标注平台对自动标注的语料进行校正, 形成700份标注病历数据集;
(2) 基于700份标注病历, 按照4:1的比例将数据集分为560份训练集和140份测试集, 采用Pipeline方式对病历数据进行分词、词性标注和命名实体识别;
(3) 对比增加数据集前后的实验结果, 评估本研究所提方法在原始300份标注病历以及增加标注数据后的700份病历上的性能。
4.3 数据处理
基于原始已标注的300份训练集, 按照4:1的比例分为240份训练集和60份测试集。5类医疗实体的统计数据如表1所示。
表1 5类医疗实体的统计数据
| 数据集 | 症状和体征 | 检查和 检验 | 治疗 | 疾病和 诊断 | 身体部位 | 合计 |
|---|---|---|---|---|---|---|
| 训练集 | 6 486 | 7 987 | 853 | 515 | 8 942 | 24 783 |
| 测试集 | 1 345 | 1 559 | 195 | 207 | 1 777 | 5 083 |
基于BEMS序列标注方法对医疗实体进行编码, 如表2所示。
表2 BEMS编码
| 序号 | 编码 | 实体类别 |
|---|---|---|
| 1 | B-SYMPTOM | 症状和体征 |
| 2 | E-SYMPTOM | |
| 3 | M-SYMPTOM | |
| 4 | S-SYMPTOM | |
| 5 | B-CHECK | 检查和检验 |
| 6 | E-CHECK | |
| 7 | M-CHECK | |
| 8 | S-CHECK | |
| 9 | B-TREATMENT | 治疗 |
| 10 | E-TREATMENT | |
| 11 | M-TREATMENT | |
| 12 | S-TREATMENT | |
| 13 | B-DISEASE | 疾病和诊断 |
| 14 | E-DISEASE | |
| 15 | M-DISEASE | |
| 16 | S-DISEASE | |
| 17 | B-BODY | 身体部位 |
| 18 | E-BODY | |
| 19 | M-BODY | |
| 20 | S-BODY | |
| 21 | O | 非医疗实体 |
4.4 实验结果
(1) 基于单字特征的CRF模型
表3 训练集实验结果
| 症状和 体征 | 检查和 检验 | 治疗 | 疾病和 诊断 | 身体 部位 | 总体 | |
|---|---|---|---|---|---|---|
| P | 0.9898 | 0.9554 | 0.9588 | 0.9703 | 0.9237 | 0.9531 |
| R | 0.9864 | 0.8233 | 0.9555 | 0.9515 | 0.9358 | 0.9138 |
| F值 | 0.9881 | 0.8845 | 0.9571 | 0.9608 | 0.9297 | 0.9331 |
表4 测试集实验结果
| 症状和体征 | 检查和检验 | 治疗 | 疾病和诊断 | 身体 部位 | 总体 | |
|---|---|---|---|---|---|---|
| P | 0.9439 | 0.9091 | 0.7945 | 0.7772 | 0.8419 | 0.8860 |
| R | 0.9636 | 0.7505 | 0.5949 | 0.6908 | 0.8149 | 0.8210 |
| F值 | 0.9536 | 0.8222 | 0.6804 | 0.7315 | 0.8281 | 0.8522 |
(2) 基于词的Pipeline处理
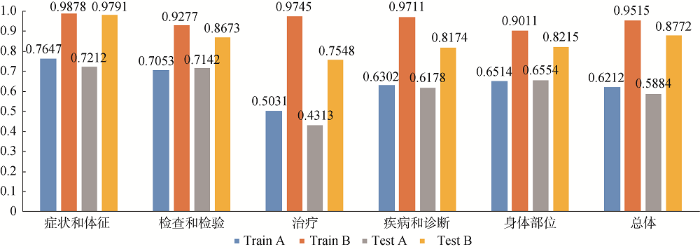
采用Pipeline方式进行实验, 比较补充数据集前后的F值, 实验结果如图6所示。Train A和Test A分别表示未增加标注数据时在训练集和测试集上的表现, Train B和Test B分别表示增加标注数据后在训练集和测试集上的表现。实验表明, 基于300份病历数据, 训练集和测试集上的F值均低于0.8。因此, 基于词的临床命名实体识别方法在较少的语料上未能达到理想效果。通过本文构建的标注平台, 得到标注病历700份, 在测试集上的总体F值可达0.8772, 较基于原始标注病历数据集的F值0.5884提升32.9%, 从而验证了本研究所提标注方法的可行性。
图6
4.5 讨论与总结
医疗实体, 如症状和体征、检查和检验、治疗、疾病和诊断、身体部位等, 均具有明确的词性特征, 因此, 通过加入词性等特征可增加信息量, 在分词准确的前提下, 基于词的方法必然有利于提高医疗实体识别准确率, 这对于提升临床文本分析与处理能力并进一步促进医疗大数据背景下的临床知识发现具有积极的推动作用。然而, 从实验结果可以看出, 在小数据集上, 基于单字特征的标注方法即可取得较理想的效果, 而基于词的Pipeline方法拟合效果反而不理想。通过扩充数据集, 当增加400份标注数据后, 基于词的方法性能得到明显提升, 在验证Pipeline方法有效的同时, 也说明数据集的大小是影响算法性能的直接因素之一。
本文针对中文电子病历开展标注方法研究。在实验部分通过基于单字特征的CRF方法训练模型, 经过性能评估, 医疗实体识别效果较好; 因此, 利用基于单字特征得到的模型, 训练未标注的病历数据, 并基于本研究构建的中文电子病历标注平台, 对自动标注数据进行审校, 从而扩充病历标注语料集; 在此基础上, 进行分词、词性标注和临床命名实体识别, 最后基于词的Pipeline方法取得较好的医疗实体识别效果。
5 结语
医疗大数据为医学知识发现带来了空前的机遇。随着医疗信息系统的普及以及信息技术的高速发展, 医疗数据将呈现更大规模, 且结构更为复杂, 这给医疗大数据处理提出更大挑战; 与此同时, 医疗大数据带来的社会和经济效益将更加显著, 临床文本处理与知识发现研究将在医学领域发挥更大作用。
针对大数据时代中文临床文本处理与知识发现的实际需求, 本文针对中文电子病历设计标注流程并构建标注平台, 基于评测语料对中文电子病历标注方法进行实证研究。实验表明, 通过本研究构建的标注平台可实现对标注病历语料的有效扩充, 且基于词的Pipeline方法可得到较好的临床命名实体识别性能, 从而验证了病历标注方法的可行性, 为临床知识发现相关研究提供方法参考。未来的研究工作将围绕以下方向开展。
(1) 加强基于电子病历的临床语料库建设
语料库是开展知识发现的研究基础。虽然临床自然语言处理日益受到关注, 然而公开可用的中文临床文本语料库尚未形成。需要加快对临床文本的规范化处理进程, 以病程记录等临床文本为研究对象, 加强基于电子病历的临床语料库建设。规范化是临床知识共享与服务的必要前提, 也是医疗大数据整合与利用的重要手段, 因此, 有必要制定科学有效的语料库建设指南, 不断优化临床文本的规范化流程, 并加强智能化手段在临床文本自动化分析与处理中的运用, 提高数据质量并改善知识发现的效果。
(2) 推进临床知识发现工具的研发工作
以面向知识发现的中文电子病历标注平台构建为契机, 借鉴cTAKES在临床文本自然语言处理方面的系统建设经验, 持续推进临床知识发现工具的研发工作。大数据时代, 数据和需求均随着时间的推移而动态发展, 可借鉴UIMA非结构化信息管理架构思想, 针对中文临床文本特点和健康医疗实际需求, 围绕知识发现的全生命周期, 研发临床文本处理与知识发现系列组件, 为基于医疗大数据的知识发现提供灵活高效的工具和平台。
(3) 注重对人机交互知识发现方法的研究
人机交互是知识发现的研究趋势。以文本标注为例, 标注的思想是以计算机可理解的方式对人类知识进行建模, 以帮助计算机处理非结构化信息。该项工作的复杂性在很大程度上取决于语言、领域和文本质量。通过人机交互的知识发现方法, 将人类智慧纳入知识发现的循环之中, 有助于提高知识发现的质量。目前, NLPReViz[35]基于交互式机器学习、信息可视化和自然语言处理方法, 为临床医生和研究人员提供了训练、审查和修订NLP模型的原型工具。
(4) 逐步完善临床知识管理与服务体系
为实现知识的高效利用和优质服务, 必须对知识进行科学有效的管理。梅奥诊所已成功将知识管理方法用于指导临床指南更新[36], 为知识资源的互操作提供了方法借鉴。知识共享是避免资源重复和浪费的有效手段, 且更为重要的是, 知识共享有利于形成各种知识资产链, 以提供更高质量的临床服务。此外, 已发现的知识是新知识发现的基础, 知识的共享与利用将推动医学创新。需要指出的是, 临床知识管理与服务尚面临诸多问题亟待解决, 例如临床知识的版权和知识产权归属问题、数据安全性和机密性问题以及缺乏对失败型知识的分享等。随着临床知识管理与服务体系的逐步完善, 基于医疗大数据的知识发现与创新将促进健康医疗服务向着更深更广的方向发展。
支撑数据
支撑数据由作者自存储, E-mail: zhao.wanqing@imicams.ac.cn。
[1] 赵琬清. emr_original.zip. 电子病历原始数据.
[2] 赵琬清. emr_crf.py, emr_assessment.py. 算法实现程序代码.
[3] 赵琬清. result_data.txt. 实验结果数据.
利益冲突声明
所有作者声明不存在利益冲突关系。
作者贡献声明
胡佳慧: 设计研究方案, 撰写论文;
方安: 提出研究思路, 修改论文;
赵琬清: 数据获取, 实验验证;
杨晨柳: 资料收集, 数据标注;
任慧玲: 标注数据审校。
参考文献
Automatic Identification of Substance Abuse from Social History in Clinical Text
[C]//
Predicting Chronic Disease Hospitalizations from Electronic Health Records: An Interpretable Classification Approach
[J].
Mining Electronic Health Records Data: Domestic Violence and Adverse Health Effects
[J].
Role of Medicines of Unknown Identity in Adverse Drug Reaction-Related Hospitalizations in Developing Countries: Evidence from a Cross-Sectional Study in a Teaching Hospital in the Lao People’s Democratic Republic
[J].
Automatic Recognition of Disorders, Findings, Pharmaceuticals and Body Structures from Clinical Text: An Annotation and Machine Learning Study
[J].
Big Data in Health Care: Using Analytics to Identify and Manage High-Risk and High-Cost Patients
[J].
Text Mining for Traditional Chinese Medical Knowledge Discovery: A Survey
[J].
Challenges in Clinical Natural Language Processing for Automated Disorder Normalization
[J].
电子病历命名实体识别和实体关系抽取研究综述
[J].
DOI:10.3724/SP.J.1004.2014.01537
URL
Magsci
[本文引用: 2]

<p>电子病历(Electronic medical records,EMR)产生于临床治疗过程,其中命名实体和实体关系反映了患者健康状况,包含了大量与患者健康状况密切相关的医疗知识,因而对它们的识别和抽取是信息抽取研究在医疗领域的重要扩展. 本文首先讨论了电子病历文本的语言特点和结构特点,然后在梳理了命名实体识别和实体关系抽取研究一般思路的基础上,分析了电子病历命名实体识别、实体修饰识别和实体关系抽取研究的具体任务和对应任务的主要研究方法. 本文还介绍了相关的共享评测任务和标注语料库以及医疗领域几个重要的词典和知识库等资源. 最后对这一研究领域仍需解决的问题和未来的发展方向作了展望.</p>
An Overview of Research on Electronic Medical Record Oriented Named Entity Recognition and Entity Relation Extraction
[J].
DOI:10.3724/SP.J.1004.2014.01537
URL
Magsci
[本文引用: 2]

<p>电子病历(Electronic medical records,EMR)产生于临床治疗过程,其中命名实体和实体关系反映了患者健康状况,包含了大量与患者健康状况密切相关的医疗知识,因而对它们的识别和抽取是信息抽取研究在医疗领域的重要扩展. 本文首先讨论了电子病历文本的语言特点和结构特点,然后在梳理了命名实体识别和实体关系抽取研究一般思路的基础上,分析了电子病历命名实体识别、实体修饰识别和实体关系抽取研究的具体任务和对应任务的主要研究方法. 本文还介绍了相关的共享评测任务和标注语料库以及医疗领域几个重要的词典和知识库等资源. 最后对这一研究领域仍需解决的问题和未来的发展方向作了展望.</p>
基于多维度聚合的网络资源知识发现框架研究
[J].本文以网络资源为研究对象, 以多维度聚合为主要手段, 针对网络资源内容的大数据化、 动态化、 多维度等特征, 探索基于语义关联的网络资源深度揭示与多维度聚合, 以此为基础研究基于多维度聚合的网络资源知识发现框架, 进而研发基于多维度聚合的网络资源知识发现技术系统, 并结合特定领域、 特定需求进行应用示范与对策研究。
Research on Knowledge Discovery Framework of Internet Resource Based on Multi-Dimensional Aggregation
[J].本文以网络资源为研究对象, 以多维度聚合为主要手段, 针对网络资源内容的大数据化、 动态化、 多维度等特征, 探索基于语义关联的网络资源深度揭示与多维度聚合, 以此为基础研究基于多维度聚合的网络资源知识发现框架, 进而研发基于多维度聚合的网络资源知识发现技术系统, 并结合特定领域、 特定需求进行应用示范与对策研究。
面向科技文献的语义检索系统研究综述
[J].
Review on Semantic Retrieval System for Scientific Literature
[J].
知识服务环境下语义化开放接口应用研究
[J].
Research on the Application of Semantic Open Interface Under Knowledge Service Environment
[J].
Data Age 2025: The Evolution of Data to Life-Critical Don’t Focus on Big Data; Focus on Data That’s Big[R]
SIGLEX. SemEval-2018: International Workshop on Semantic Evaluation
[EB/OL]. [
i2b2 tranSMART Foundation. i2b2: Informatics for Integrating Biology & the Bedside
[EB/OL].[2018-06-19]. .
Text Analytics for Life Science Using the Unstructured Information Management Architecture
[J].
Mayo Clinical Text Analysis and Knowledge Extraction System (cTAKES): Architecture, Component Evaluation and Applications
[J].
DeepPhe: A Natural Language Processing System for Extracting Cancer Phenotypes from Clinical Records
[J].
Alphabetical List of Part-of-Speech Tags Used in the Penn Treebank Project
[EB/OL]. [
The Unified Medical Language System (UMLS): Integrating Biomedical Terminology
[J].
Capturing the Patient’s Perspective: A Review of Advances in Natural Language Processing of Health-Related Text
[J].
2010 i2b2/VA Challenge on Concepts, Assertions, and Relations in Clinical Text
[J].
中文电子病历命名实体和实体关系语料库构建
[J].
Corpus Construction for Named Entities and Entity Relations on Chinese Electronic Medical Records
[J].
Building a Comprehensive Syntactic and Semantic Corpus of Chinese Clinical Texts
[J].
中文电子病历命名实体标注语料库构建
[J].
The Construction of Annotated Corpora of Named Entities for Chinese Electronic Medical Records
[J].
Iterative Development of Family History Annotation Guidelines Using a Synthetic Corpus of Clinical Text
[C]//
Language, Knowledge, and Intelligence
[C]//
CCKS 2018: China Conference on Knowledge Graph and Semantic Computing
[EB/OL]. [
CHIP 2018: The 4th China Conference on Health Information Processing
[EB/OL]. [
Conditional Random Fields: Probabilistic Models for Segmenting and Labeling Sequence Data
[C]//
Natural Language Annotation for Machine Learning: A Guide to Corpus-Building for Applications
[M].
NLPReViz: An Interactive Tool for Natural Language Processing on Clinical Text
[J].
Knowledge as a Service at the Point of Care
[J].