第43次《中国互联网络发展状况统计报告》
1
2019
... 2019年2月28日, 中国互联网络中心(CNNIC)发布的第43次中国互联网发展状况统计报告显示, 截至2018年12月, 我国网民规模达8.29亿, 较2017年底增加3.8%[1].在大数据时代, 越来越多的人们倾向借助互联网进行信息的检索和交换, 尤其在医疗领域, 在线预约挂号、在线问诊、远程医疗等应用大大提高了就医效率, 在一定程度上缓解了医疗资源分配不均衡、不充分的问题[2].与传统的以医生为中心的医疗信息服务不同, 如今的互联网健康社区具有高度的社交互动性和参与度, 提供了医生与患者和患者之间的信息交流途径[3].为了更好地聆听患者的声音, 提供以患者为中心的服务, 面对爆炸式增长的互联网患者提问数据, 如何进行信息提取和有效利用成为重要的研究方向. ...
第43次《中国互联网络发展状况统计报告》
1
2019
... 2019年2月28日, 中国互联网络中心(CNNIC)发布的第43次中国互联网发展状况统计报告显示, 截至2018年12月, 我国网民规模达8.29亿, 较2017年底增加3.8%[1].在大数据时代, 越来越多的人们倾向借助互联网进行信息的检索和交换, 尤其在医疗领域, 在线预约挂号、在线问诊、远程医疗等应用大大提高了就医效率, 在一定程度上缓解了医疗资源分配不均衡、不充分的问题[2].与传统的以医生为中心的医疗信息服务不同, 如今的互联网健康社区具有高度的社交互动性和参与度, 提供了医生与患者和患者之间的信息交流途径[3].为了更好地聆听患者的声音, 提供以患者为中心的服务, 面对爆炸式增长的互联网患者提问数据, 如何进行信息提取和有效利用成为重要的研究方向. ...
The Creation of Social Value: Can an Online Health Community Reduce Rural-urban Health Disparities?
1
2016
... 2019年2月28日, 中国互联网络中心(CNNIC)发布的第43次中国互联网发展状况统计报告显示, 截至2018年12月, 我国网民规模达8.29亿, 较2017年底增加3.8%[1].在大数据时代, 越来越多的人们倾向借助互联网进行信息的检索和交换, 尤其在医疗领域, 在线预约挂号、在线问诊、远程医疗等应用大大提高了就医效率, 在一定程度上缓解了医疗资源分配不均衡、不充分的问题[2].与传统的以医生为中心的医疗信息服务不同, 如今的互联网健康社区具有高度的社交互动性和参与度, 提供了医生与患者和患者之间的信息交流途径[3].为了更好地聆听患者的声音, 提供以患者为中心的服务, 面对爆炸式增长的互联网患者提问数据, 如何进行信息提取和有效利用成为重要的研究方向. ...
A New Dimension of Health Care: Systematic Review of the Uses, Benefits, and Limitations of Social Media for Health Communication
1
2013
... 2019年2月28日, 中国互联网络中心(CNNIC)发布的第43次中国互联网发展状况统计报告显示, 截至2018年12月, 我国网民规模达8.29亿, 较2017年底增加3.8%[1].在大数据时代, 越来越多的人们倾向借助互联网进行信息的检索和交换, 尤其在医疗领域, 在线预约挂号、在线问诊、远程医疗等应用大大提高了就医效率, 在一定程度上缓解了医疗资源分配不均衡、不充分的问题[2].与传统的以医生为中心的医疗信息服务不同, 如今的互联网健康社区具有高度的社交互动性和参与度, 提供了医生与患者和患者之间的信息交流途径[3].为了更好地聆听患者的声音, 提供以患者为中心的服务, 面对爆炸式增长的互联网患者提问数据, 如何进行信息提取和有效利用成为重要的研究方向. ...
序列标注模型中的字粒度特征提取方案研究——以CCKS2017:Task2临床病历命名实体识别任务为例
1
2018
... 作为信息抽取的子任务, 命名实体识别(Named Entity Recognition, NER)方法可从非结构化文本中, 抽取蛋白质、疾病名、药物名等实体[4,5].而互联网患者提问文本具有噪声大、用词不规范等特点, 为实体识别工作带来了一定挑战.另外, 现有命名实体识别研究大多基于传统的统计机器学习方法, 需要训练数据与测试数据具有相同分布, 且对数据量有一定要求, 尤其当研究数据具有较强时效性时, 需要大量人工标注的传统机器学习方法往往不再适用. ...
序列标注模型中的字粒度特征提取方案研究——以CCKS2017:Task2临床病历命名实体识别任务为例
1
2018
... 作为信息抽取的子任务, 命名实体识别(Named Entity Recognition, NER)方法可从非结构化文本中, 抽取蛋白质、疾病名、药物名等实体[4,5].而互联网患者提问文本具有噪声大、用词不规范等特点, 为实体识别工作带来了一定挑战.另外, 现有命名实体识别研究大多基于传统的统计机器学习方法, 需要训练数据与测试数据具有相同分布, 且对数据量有一定要求, 尤其当研究数据具有较强时效性时, 需要大量人工标注的传统机器学习方法往往不再适用. ...
基于特征耦合泛化的药名实体识别
1
2014
... 作为信息抽取的子任务, 命名实体识别(Named Entity Recognition, NER)方法可从非结构化文本中, 抽取蛋白质、疾病名、药物名等实体[4,5].而互联网患者提问文本具有噪声大、用词不规范等特点, 为实体识别工作带来了一定挑战.另外, 现有命名实体识别研究大多基于传统的统计机器学习方法, 需要训练数据与测试数据具有相同分布, 且对数据量有一定要求, 尤其当研究数据具有较强时效性时, 需要大量人工标注的传统机器学习方法往往不再适用. ...
基于特征耦合泛化的药名实体识别
1
2014
... 作为信息抽取的子任务, 命名实体识别(Named Entity Recognition, NER)方法可从非结构化文本中, 抽取蛋白质、疾病名、药物名等实体[4,5].而互联网患者提问文本具有噪声大、用词不规范等特点, 为实体识别工作带来了一定挑战.另外, 现有命名实体识别研究大多基于传统的统计机器学习方法, 需要训练数据与测试数据具有相同分布, 且对数据量有一定要求, 尤其当研究数据具有较强时效性时, 需要大量人工标注的传统机器学习方法往往不再适用. ...
Message Understanding Conference-6: A Brief History
1
1996
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Conditional Random Fields: Probabilistic Models for Segmenting and Labeling Sequence Data
1
2001
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Nymble: A High-performance Learning Name-finder
1
1997
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Maximum Entropy Models for Named Entity Recognition
1
2003
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Learning Task-dependent Distributed Representations by Backpropagation Through Structure
1
1996
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Long Short-Term Memory
1
1997
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Framewise Phoneme Classification with Bidirectional LSTM and Other Neural Network Architectures
1
2005
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
An Overview of Named Entity Recognition
1
2018
... 命名实体识别任务是指在给定文本中确定实体边界, 并将其划分至特定类别, 如人名、地名、机构名的识别.作为自然语言处理领域的基础任务, 命名实体识别可应用于如信息提取、关系提取、问答系统等下游任务.自1996年第6届消息理解会议[6]首次提出命名实体识别任务至今, 相关技术得到了广泛的研究和发展.从早期基于统计和手工编写规则的方法, 到基于特征工程和机器学习的方法, 包括条件随机场(Conditional Random Field, CRF)[7]、隐马尔可夫模型(Hidden Markov Model, HMM)[8]、最大熵模型(Maximum Entropy, ME)[9]等, 命名实体识别的性能得到了较大提升.近年来, 随着神经网络的发展, 由于循环神经网络(Recurrent Neural Network, RNN)[10]能够有效捕捉句子的上下文信息, 尤其擅长序列标记任务, 而且基于循环神经网络改进的长短时记忆模型(Long-Short Term Memory, LSTM)[11]可以有效克服长距离依赖问题, 因此大量研究选择基于双向长短时记忆模型(Bi-directional LSTM, Bi-LSTM)[12], 根据上下文信息进行文本特征提取, 并结合条件随机场方法进行命名实体识别[13]. ...
Domain Adaptation with Structural Correspondence Learning
1
2006
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Instance Weighting for Domain Adaptation in NLP
1
2007
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Transfer Learning for Sequence Tagging with Hierarchical Recurrent Networks
1
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Boosting for Transfer Learning
2
2007
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
... (1) 基于实例的迁移学习原理是通过调节源领域和目标领域的数据权重, 借助源领域样本丰富目标任务训练数据, 实现迁移学习任务.例如, 高冰涛等[36]在隐马尔可夫模型的基础上, 利用数据引力方法评估源域样本权重, 提出基于权值的隐马尔可夫模型, 通过在GENIA语料库中对蛋白质实体的识别实验, 证明该迁移方法只需少量目标领域标注样本即可得到良好的命名实体识别性能.王红斌等[37]基于TrAdaboost算法[17]进行改进, 通过对源领域和目标领域数据权值的自动调整, 进行基于样本选择的迁移学习, 实验证明该方法能够在新兴的、缺乏标准训练语料的领域, 获得较好的命名实体识别效果.以上基于实例的迁移方法, 在一定程度上解决了领域适配和负迁移问题, 但其识别效果依赖于源领域的标注数量和质量, 当标注量不足或质量较差时, 迁移效果也会随之受到影响. ...
Transferring Naive Bayes Classifiers for Text Classification
1
2007
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Co-clustering Based Classification for Out-of-domain Documents
1
2007
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Topic-bridged PLSA for Cross-domain Text Classification
1
2008
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Domain Adaptation via Transfer Component Analysis
1
2010
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Cross Domain Distribution Adaptation via Kernel Mapping
1
2009
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
一种基于跨领域典型相关性分析的迁移学习方法
1
2015
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
一种基于跨领域典型相关性分析的迁移学习方法
1
2015
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Transfer Learning for Class Imbalance Problems with Inadequate Data
1
2016
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
A Transfer Cost-sensitive Boosting Approach for Cross-project Defect Prediction
1
2017
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Cross-domain Sentiment Classification via Spectral Feature Alignment
1
2010
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Automatically Extracting Polarity-bearing Topics for Cross-domain Sentiment Classification
1
2011
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Transitive Transfer Learning
1
2015
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
基于迁移学习微博情绪分类研究——以H7N9微博为例
1
2016
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
基于迁移学习微博情绪分类研究——以H7N9微博为例
1
2016
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Cross-domain Sentiment Classification via Topic-related TrAdaBoost
1
2017
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
基于深度循环神经网络的跨领域文本情感分析
1
2018
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
基于深度循环神经网络的跨领域文本情感分析
1
2018
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
Transfer Learning for Biomedical Named Entity Recognition with Neural Networks
3
2018
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
... (3) 基于模型的迁移学习原理是通过对领域间模型和参数的共享, 实现对目标任务的迁移.如Giorgi等[32]利用深度神经网络, 在大型有噪声的生物医学数据集进行模型的训练, 然后将参数迁移至小型目标数据集, 实验结果与最新基线模型相比, 平均错误率降低了约9%, 证明了该模型迁移方法应用于生物医学领域命名实体识别的有效性.Corbett等[33]使用GloVe[39]对生物医学相关语料进行词向量的预训练, 并与传统深度学习方法结合, 实现了基于两阶段训练的模型迁移.经实验对比, 证明了该方法可显著提升无迁移的深度学习模型效果.由于模型迁移在一定程度上降低了对目标领域标注数据量以及特征分布的要求, 且基于大量数据预训练的网络模型具有可重复利用、泛化能力强等特点, 使得模型迁移方法成为自然语言处理领域的重要研究方向. ...
... 实验数据集的划分借鉴Giorgi等[32]的研究, 将目标领域数据集中60%的样本作为训练集, 10%作为验证集, 30%用于识别结果预测的测试集. ...
Chemlistem: Chemical Named Entity Recognition Using Recurrent Neural Networks
2
2018
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
... (3) 基于模型的迁移学习原理是通过对领域间模型和参数的共享, 实现对目标任务的迁移.如Giorgi等[32]利用深度神经网络, 在大型有噪声的生物医学数据集进行模型的训练, 然后将参数迁移至小型目标数据集, 实验结果与最新基线模型相比, 平均错误率降低了约9%, 证明了该模型迁移方法应用于生物医学领域命名实体识别的有效性.Corbett等[33]使用GloVe[39]对生物医学相关语料进行词向量的预训练, 并与传统深度学习方法结合, 实现了基于两阶段训练的模型迁移.经实验对比, 证明了该方法可显著提升无迁移的深度学习模型效果.由于模型迁移在一定程度上降低了对目标领域标注数据量以及特征分布的要求, 且基于大量数据预训练的网络模型具有可重复利用、泛化能力强等特点, 使得模型迁移方法成为自然语言处理领域的重要研究方向. ...
A Survey on Concept Drift Adaptation
1
2014
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
A Survey on Transfer Learning
2
2009
... 在自然语言处理领域, 迁移学习方法主要应用于跨领域序列标注[14,15,16]、文本分类[17,18,19,20,21,22,23,24,25]、情感分类[26,27,28,29,30,31]、命名实体识别[32,33]等任务.由于领域间的数据分布存在差异, 在实际迁移过程中往往面临概念漂移[34]和负迁移[35]问题.为解决以上问题, 现有的跨领域命名实体识别迁移学习方法主要从基于实例、特征和模型的角度展开研究. ...
... (2) 基于特征的迁移学习是指通过特征变换, 找到一个“好的”特征表示, 减少领域间的差异, 以提高模型的性能[35].例如, Pan等[38]提出迁移联合嵌入(Transfer Joint Embedding, TJE)模型, 首先将不同领域的高维特征和标签映射到一个统一的低维空间, 然后在该低维空间中, 利用最近邻方法实现跨领域命名实体识别.但基于特征选择的迁移方法依赖于各领域数据的分布情况, 若数据分布过于稀疏, 则难以建立适宜的特征空间, 从而影响模型性能. ...
BioTrHMM:基于迁移学习的生物医学命名实体识别算法
1
2019
... (1) 基于实例的迁移学习原理是通过调节源领域和目标领域的数据权重, 借助源领域样本丰富目标任务训练数据, 实现迁移学习任务.例如, 高冰涛等[36]在隐马尔可夫模型的基础上, 利用数据引力方法评估源域样本权重, 提出基于权值的隐马尔可夫模型, 通过在GENIA语料库中对蛋白质实体的识别实验, 证明该迁移方法只需少量目标领域标注样本即可得到良好的命名实体识别性能.王红斌等[37]基于TrAdaboost算法[17]进行改进, 通过对源领域和目标领域数据权值的自动调整, 进行基于样本选择的迁移学习, 实验证明该方法能够在新兴的、缺乏标准训练语料的领域, 获得较好的命名实体识别效果.以上基于实例的迁移方法, 在一定程度上解决了领域适配和负迁移问题, 但其识别效果依赖于源领域的标注数量和质量, 当标注量不足或质量较差时, 迁移效果也会随之受到影响. ...
BioTrHMM:基于迁移学习的生物医学命名实体识别算法
1
2019
... (1) 基于实例的迁移学习原理是通过调节源领域和目标领域的数据权重, 借助源领域样本丰富目标任务训练数据, 实现迁移学习任务.例如, 高冰涛等[36]在隐马尔可夫模型的基础上, 利用数据引力方法评估源域样本权重, 提出基于权值的隐马尔可夫模型, 通过在GENIA语料库中对蛋白质实体的识别实验, 证明该迁移方法只需少量目标领域标注样本即可得到良好的命名实体识别性能.王红斌等[37]基于TrAdaboost算法[17]进行改进, 通过对源领域和目标领域数据权值的自动调整, 进行基于样本选择的迁移学习, 实验证明该方法能够在新兴的、缺乏标准训练语料的领域, 获得较好的命名实体识别效果.以上基于实例的迁移方法, 在一定程度上解决了领域适配和负迁移问题, 但其识别效果依赖于源领域的标注数量和质量, 当标注量不足或质量较差时, 迁移效果也会随之受到影响. ...
融合迁移学习的中文命名实体识别
1
2017
... (1) 基于实例的迁移学习原理是通过调节源领域和目标领域的数据权重, 借助源领域样本丰富目标任务训练数据, 实现迁移学习任务.例如, 高冰涛等[36]在隐马尔可夫模型的基础上, 利用数据引力方法评估源域样本权重, 提出基于权值的隐马尔可夫模型, 通过在GENIA语料库中对蛋白质实体的识别实验, 证明该迁移方法只需少量目标领域标注样本即可得到良好的命名实体识别性能.王红斌等[37]基于TrAdaboost算法[17]进行改进, 通过对源领域和目标领域数据权值的自动调整, 进行基于样本选择的迁移学习, 实验证明该方法能够在新兴的、缺乏标准训练语料的领域, 获得较好的命名实体识别效果.以上基于实例的迁移方法, 在一定程度上解决了领域适配和负迁移问题, 但其识别效果依赖于源领域的标注数量和质量, 当标注量不足或质量较差时, 迁移效果也会随之受到影响. ...
融合迁移学习的中文命名实体识别
1
2017
... (1) 基于实例的迁移学习原理是通过调节源领域和目标领域的数据权重, 借助源领域样本丰富目标任务训练数据, 实现迁移学习任务.例如, 高冰涛等[36]在隐马尔可夫模型的基础上, 利用数据引力方法评估源域样本权重, 提出基于权值的隐马尔可夫模型, 通过在GENIA语料库中对蛋白质实体的识别实验, 证明该迁移方法只需少量目标领域标注样本即可得到良好的命名实体识别性能.王红斌等[37]基于TrAdaboost算法[17]进行改进, 通过对源领域和目标领域数据权值的自动调整, 进行基于样本选择的迁移学习, 实验证明该方法能够在新兴的、缺乏标准训练语料的领域, 获得较好的命名实体识别效果.以上基于实例的迁移方法, 在一定程度上解决了领域适配和负迁移问题, 但其识别效果依赖于源领域的标注数量和质量, 当标注量不足或质量较差时, 迁移效果也会随之受到影响. ...
Transfer Joint Embedding for Cross-Domain Named Entity Recognition
1
2013
... (2) 基于特征的迁移学习是指通过特征变换, 找到一个“好的”特征表示, 减少领域间的差异, 以提高模型的性能[35].例如, Pan等[38]提出迁移联合嵌入(Transfer Joint Embedding, TJE)模型, 首先将不同领域的高维特征和标签映射到一个统一的低维空间, 然后在该低维空间中, 利用最近邻方法实现跨领域命名实体识别.但基于特征选择的迁移方法依赖于各领域数据的分布情况, 若数据分布过于稀疏, 则难以建立适宜的特征空间, 从而影响模型性能. ...
GloVe: Global Vectors for Word Representation
1
2014
... (3) 基于模型的迁移学习原理是通过对领域间模型和参数的共享, 实现对目标任务的迁移.如Giorgi等[32]利用深度神经网络, 在大型有噪声的生物医学数据集进行模型的训练, 然后将参数迁移至小型目标数据集, 实验结果与最新基线模型相比, 平均错误率降低了约9%, 证明了该模型迁移方法应用于生物医学领域命名实体识别的有效性.Corbett等[33]使用GloVe[39]对生物医学相关语料进行词向量的预训练, 并与传统深度学习方法结合, 实现了基于两阶段训练的模型迁移.经实验对比, 证明了该方法可显著提升无迁移的深度学习模型效果.由于模型迁移在一定程度上降低了对目标领域标注数据量以及特征分布的要求, 且基于大量数据预训练的网络模型具有可重复利用、泛化能力强等特点, 使得模型迁移方法成为自然语言处理领域的重要研究方向. ...
Bert: Pre-training of Deep Bidirectional Transformers for Language Understanding
1
... 2018年10月, 谷歌发布了自然语言处理领域的BERT语言模型[40], 凭借其强大的性能刷新了11项自然语言处理任务记录.作为首个在模型所有层中实现双向训练的语言模型, BERT融合了ELMo[41]和OpenAI GPT[42]的优点.以BERT-Base为例, 模型采用两阶段的训练方式, 首先利用Transformer编码器进行特征抽取, 通过遮蔽语言模型(Masked Language Model, MLM)与下一句预测(Next Sentence Prediction, NSP)相结合的训练方法, 对中文维基百科语料进行第一阶段的预训练, 然后允许针对不同的目标任务, 在预训练模型的基础上进行特征集成(Feature-based)或微调(Fine-tuning). ...
Deep Contextualized Word Representations
1
... 2018年10月, 谷歌发布了自然语言处理领域的BERT语言模型[40], 凭借其强大的性能刷新了11项自然语言处理任务记录.作为首个在模型所有层中实现双向训练的语言模型, BERT融合了ELMo[41]和OpenAI GPT[42]的优点.以BERT-Base为例, 模型采用两阶段的训练方式, 首先利用Transformer编码器进行特征抽取, 通过遮蔽语言模型(Masked Language Model, MLM)与下一句预测(Next Sentence Prediction, NSP)相结合的训练方法, 对中文维基百科语料进行第一阶段的预训练, 然后允许针对不同的目标任务, 在预训练模型的基础上进行特征集成(Feature-based)或微调(Fine-tuning). ...
Improving Language Understanding by Generative Pre-training
1
2019
... 2018年10月, 谷歌发布了自然语言处理领域的BERT语言模型[40], 凭借其强大的性能刷新了11项自然语言处理任务记录.作为首个在模型所有层中实现双向训练的语言模型, BERT融合了ELMo[41]和OpenAI GPT[42]的优点.以BERT-Base为例, 模型采用两阶段的训练方式, 首先利用Transformer编码器进行特征抽取, 通过遮蔽语言模型(Masked Language Model, MLM)与下一句预测(Next Sentence Prediction, NSP)相结合的训练方法, 对中文维基百科语料进行第一阶段的预训练, 然后允许针对不同的目标任务, 在预训练模型的基础上进行特征集成(Feature-based)或微调(Fine-tuning). ...
Enhancing Clinical Concept Extraction with Contextual Embedding
1
... 在实际应用中, 经过大量数据预训练的网络结构可以为目标学习任务提供丰富的先验知识, 节省训练大型神经网络的时间成本, 提高模型的泛化能力和鲁棒性, 同时还能节省大量的人工标注工作, 有利于更好地处理小数据问题和个性化问题.例如, 针对生物医学领域的文本挖掘任务, Si等[43]通过比较传统词嵌入方法Word2Vec、GloVe、fastText与上下文嵌入BERT、ELMo方法在临床文本中的概念提取能力, 展示了上下文嵌入方法的强大性能.Lee等[44]在BERT模型的基础上加入PubMed摘要以及PubMed Central全文进行额外的预训练, 提出一种针对临床文本的深度表示模型BioBERT, 在实体识别、关系提取、问答系统等任务中, 取得了优秀的性能. ...
Biobert: Pre-trained Biomedical Language Representation Model for Biomedical Text Mining
1
... 在实际应用中, 经过大量数据预训练的网络结构可以为目标学习任务提供丰富的先验知识, 节省训练大型神经网络的时间成本, 提高模型的泛化能力和鲁棒性, 同时还能节省大量的人工标注工作, 有利于更好地处理小数据问题和个性化问题.例如, 针对生物医学领域的文本挖掘任务, Si等[43]通过比较传统词嵌入方法Word2Vec、GloVe、fastText与上下文嵌入BERT、ELMo方法在临床文本中的概念提取能力, 展示了上下文嵌入方法的强大性能.Lee等[44]在BERT模型的基础上加入PubMed摘要以及PubMed Central全文进行额外的预训练, 提出一种针对临床文本的深度表示模型BioBERT, 在实体识别、关系提取、问答系统等任务中, 取得了优秀的性能. ...
Distributed Representations of Sentences and Documents
1
2014
... 为体现文本语义信息, 将分词结果转化成文档向量(Doc2Vec)[45], 然后利用K近邻(k-Nearest Neighbor, KNN)[46]方法, 针对每一个目标领域文档向量, 基于欧氏距离选择源领域中与其最为相似的k个样本, 加入实例迁移扩展集.最后, 经适当的k值选取, 实现基于样本选择的实例迁移过程. ...
Nearest Neighbor Pattern Classification
1
1967
... 为体现文本语义信息, 将分词结果转化成文档向量(Doc2Vec)[45], 然后利用K近邻(k-Nearest Neighbor, KNN)[46]方法, 针对每一个目标领域文档向量, 基于欧氏距离选择源领域中与其最为相似的k个样本, 加入实例迁移扩展集.最后, 经适当的k值选取, 实现基于样本选择的实例迁移过程. ...
健康领域中文自动问答的问题解析研究——以肺癌为例
1
2019
... 本文采用赵冬[47]提供的肺癌患者在线提问文本作为源领域数据集, 共计11 822条.该数据集来自寻医问药和有问必答网肺癌社区中的真实用户提问, 已详细标注了实体名称以及实体关系.为保证迁移质量, 目标领域数据同样从以上网站获取, 经过去除重复数据及无关数据, 得到共2 000条肝癌患者提问文本.各领域数据集句长分布如图4所示. ...
健康领域中文自动问答的问题解析研究——以肺癌为例
1
2019
... 本文采用赵冬[47]提供的肺癌患者在线提问文本作为源领域数据集, 共计11 822条.该数据集来自寻医问药和有问必答网肺癌社区中的真实用户提问, 已详细标注了实体名称以及实体关系.为保证迁移质量, 目标领域数据同样从以上网站获取, 经过去除重复数据及无关数据, 得到共2 000条肝癌患者提问文本.各领域数据集句长分布如图4所示. ...
Semantic Annotation of Consumer Health Questions
1
2018
... 根据本文研究目的, 结合Kilicoglu等[48]针对消费者健康问题提出的17种生物医学命名实体, 通过咨询相关专家, 实验选择9种与癌症临床诊断密切相关的标签.标注工作中使用的命名实体目录如表1所示. ...
Agreement, the F-measure, and Reliability in Information Retrieval
1
2005
... 根据以上9种实体标签的选择, 实验采用{B, I, O}标注体系, 由两名经过培训的标注人员对2 000条肝癌患者提问文本进行标注, 标注一致性检验达到较高水平, F值[49]为92%.实验中各领域数据集组成如表2所示. ...
Introduction to the CoNLL-2003 Shared Task: Language-Independent Named Entity Recognition
1
2003
... 实验评价指标借鉴CoNLL任务[50], 采用准确率(Precision, P)、召回率(Recall, R)和F1值(F1-measure, F)进行命名实体识别的效果评价.仅当整个实体的预测标签与该实体标签完全匹配时, 说明实体预测正确. ...
反馈式K近邻语义迁移学习的领域命名实体识别
1
2019
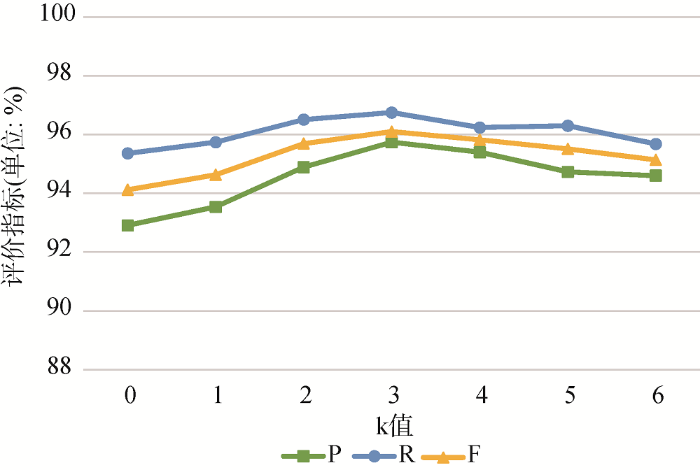
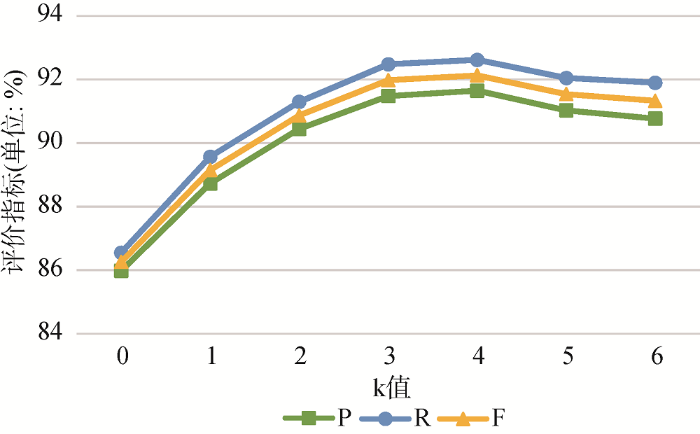
... 本文参考文献[51]进行k值实验范围的选择.因本文源领域样本数约为目标领域样本数的6倍, 设置k的实验范围为0-6, k=0表示扩展集中未添加源领域样例, k=6意味着在极端情况下, 扩展集中添加了所有源领域样例.根据不同的k值选取, KNN-BERT- BiLSTM-CRF与KNN-Word2Vec-BiLSTM-CRF模型的实例迁移对比实验结果如表5所示. ...
反馈式K近邻语义迁移学习的领域命名实体识别
1
2019
... 本文参考文献[51]进行k值实验范围的选择.因本文源领域样本数约为目标领域样本数的6倍, 设置k的实验范围为0-6, k=0表示扩展集中未添加源领域样例, k=6意味着在极端情况下, 扩展集中添加了所有源领域样例.根据不同的k值选取, KNN-BERT- BiLSTM-CRF与KNN-Word2Vec-BiLSTM-CRF模型的实例迁移对比实验结果如表5所示. ...